जनसंख्या आनुवांशिकी
इस पृथ्वी पर जीवों का महत्वपूर्ण लक्षण अनुकूलता होती है कियोंकि इनके द्वारा ही जिव इस पृथ्वी पर जिंदा रह पा रहा है चार मुख्य विकासमूलक प्रक्रियाओं: प्राकृतिक चयन, आनुवांशिक झुकाव, उत्परिवर्तन और जीन-प्रवाह के प्रभाव में युग्म-विकल्पी आवृत्ति वितरण और परिवर्तन का अध्ययन जनसंख्या आनुवांशिकी कहलाता है। इसमें जनसंख्या उप-विभाजन तथा जनसंख्या संरचना के कारकों पर भी ध्यान दिया जाता है। यह अनुकूलन और प्रजातिकरण (speciation) जैसी अवधारणाओं की व्याख्या करने का भी प्रयास करती है।
जनसंख्या आनुवांशिकी आधुनिक विकासमूलक संश्लेषण के उदभव का एक आवश्यक घटक थी। इसके प्रमुख संस्थापक सीवॉल राइट (Sewall Wright), जे. बी. एस. हाल्डेन (J. B. S. Haldane) और आर. ए. फिशर (R. A. Fisher) थे, जिन्होंने इससे संबंधित शाखा मात्रात्मक आनुवांशिकी की नींव भी रखी.
बुनियादी तत्व[संपादित करें]
जनसंख्या आनुवांशिकी जनसंख्याओं की जेनेटिक बनावट पर तथा इस बात के अध्ययन पर ध्यान केंद्रित करती है कि यह बनावट समय के साथ किस प्रकार बदलती है। जनसंख्या जीवों का एक ऐसा समुच्चय होती है, जिसमें सदस्यों का कोई भी समूह आपस में संतानोत्पत्ति कर सकता है। इसमें यह अंतर्निहित है कि सभी सदस्य एक ही प्रजाति के होते हैं तथा एक दूसरे के आस-पास ही निवास करते हैं।[1]
उदाहरण के लिये, किसी जंगल में रहने वाले एक ही प्रजाति के सभी पतंगे एक जनसंख्या हैं। इस जनसंख्या में उपस्थित किसी जीन के विभिन्न वैकल्पिक रूप हो सकते हैं, जो जीवों के फेनोटाइप (phenotypes) के बीच विविधताओं को स्पष्ट करता है। इसका एक उदाहरण पतंगों में रंग के निर्धारण के लिये उत्तरदायी एक जीन हो सकता है, जिसमें दो युग्म-विकल्पी होते हैं: काला और सफेद. किसी एकल जनसंख्या में एक जीन के लिये युग्म विकल्पियों का संपूर्ण समुच्च्य एक जीन-पूल कहलाता है; किसी युग्म विकल्पी के लिये युग्म विकल्पी आवृत्ति उस पूल के जीनों का वह भाग होती है, जो उस युग्म विकल्पी से मिलकर बने हों (उदाहरण के लिये, पतंगे में रंग निर्धारण करने वाले जीनों का कितना भाग काले युग्म-विकल्पी से मिलकर बना है). संकरण जीवों की एक जनसंख्या के अंतर्गत युग्म विकल्पियों की आवृत्तियों में परिवर्तन होने पर विकास होता है; उदाहरण के लिये, पतंगों की एक जनसंख्या में काले रंग के लिये उत्तरदायी युग्म विकल्पी अधिक आम होता जा रहा है।
किसी जनसंख्या के विकास का कारण बनने वाली कार्यविधि को समझने के लिये, इस बात पर विचार करना उपयोगी है कि किसी जनसंख्या का विकास न होने के लिये किन स्थितियों का होना आवश्यक है। हार्डी-वीनबर्ग सिद्धांत (Hardy-Weinberg principle) के अनुसार एक पर्याप्त रूप से बड़ी जनसंख्या में युग्म-विकल्पियों की आवृत्ति (जीन में विविधता) स्थिर बनी रहेगी, यदि वीर्य या अंडों के निर्माण के दौरान युग्म-विकल्पियों की यादृच्छिक अदला-बदली, तथा निषेचन के दौरान इन लिंग कोशिकाओं (sex cells) में उन युग्म-विकल्पियों का यादृच्छिक संयोजन ही उस जनसंख्या पर कार्य कर रही एकमात्र शक्ति हो। [2] ऐसी जनसंख्या को हार्डी-वीनबर्ग समतुल्यता (Hardy-Weinberg equilibrium) में माना जाता है क्योंकि इसका विकास नहीं हो रहा है।[3]
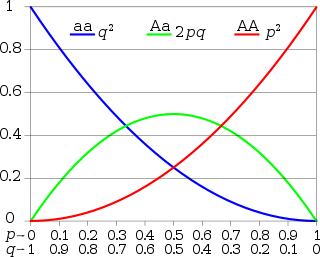
हार्डी-वीनबर्ग सिद्धांत[संपादित करें]
हार्डी-वीनबर्ग सिद्धांत (Hardy–Weinberg principle) के अनुसार किसी जनसंख्या में युग्म-विकल्पी और जेनोटाइप आवृत्तियां, दोनों ही पीढ़ी-दर-पीढ़ी स्थिर बने रहते हैं—अर्थात वे समतुल्यता की स्थिति में होते हैं—जब तक कि व्यवधान उत्पन्न करने वाले कोई विशिष्ट प्रभाव इनके संपर्क में न आएं. प्रयोगशाला के बाहर, इन “व्यवधान उत्पन्न करनेवाले प्रभावों” में से एक या अधिक सदैव प्रभावी होते हैं। हार्डी वीनबर्ग समतुल्यता प्रकृति में असंभव होती है। आनुवांशिक समतुल्यता वह आदर्श स्थिति है, जो एक आधार-रेखा प्रदान करती है, जिस पर आनुवांशिक परिवर्तन को मापा जा सके।
किसी जनसंख्या में युग्म-विकल्पियों की आवृत्तियां पीढ़ियों तक स्थिर बनीं रहती हैं, यदि निम्नलिखित शर्तों का पालन किया जा रहा हो: यादृच्छिक सहवास (random mating), कोई उत्परिवर्तन न होना (युग्म-विकल्पी परिवर्तित नहीं होते), कोई प्रवसन या उत्प्रवास न होना (जनसंख्याओं के बीच युग्म-विकल्पियों का कोई आदान-प्रदान नहीं होता), जनसंख्या का बहुत बड़ा आकार और किसी भी लक्षण के समर्थन या विरोध में कोई चयनात्मक दबाव न होना.
दो युग्म-विकल्पियों वाली किसी एकल जीन अवस्थिति के सरलतम मामले में: प्रभावशाली युग्म-विकल्पी को A द्वारा तथा अप्रभावी युग्म-विकल्पी को a द्वारा सूचित किया जाता है और उनकी आवृत्तियां (frequencies) p और q द्वारा दर्शाई जाती हैं; freq(A) = p ; freq(a) = q ; p + q = 1. यदि जनसंख्या समतुल्यता में हो, तो हमें जनसंख्या में AA समयुग्मकों (homozygotes) के लिये freq(AA) = p 2, aa समयुग्मकों के लिये freq(aa) = q 2, तथा विषमयुग्मकों (heterozygotes) के लिये freq(Aa) = 2pq प्राप्त होगी।
इन समीकरणों के आधार पर एक जनसंख्या के बारे में उपयोगी, लेकिन मापने-में-कठिन तथ्यों का निर्धारण किया जा सकता है। उदाहरण के लिये, एक मरीज की संतान उस अप्रभावी उत्परिवर्तन की वाहक होती है, जो समयुग्मक अप्रभावी बच्चों में सिस्टिक फाइब्रोसिस (cystic fibrosis) का कारण बनता है। अभिभावक यह जानना चाहती हैं कि उनके पोतों द्वारा वंशानुक्रम से इस बीमारी को ग्रहण किये जाने की कितनी संभावना है। इस प्रश्न का उत्तर देने के लिये, आनुवांशिक सलाहकार को इस संभावना की जानकारी होनी चाहिये कि वह बच्चा अप्रभावी उत्परिवर्तन के किसी वाहक के साथ सहवास करेगा। संभव है कि यह तथ्य ज्ञात न हो, परंतु बीमारी की आवृत्ति ज्ञात हो। हम जानते हैं कि यह बीमारी समयुग्मक अप्रभावी जेनोटाइप के कारण होती है; अतः हम बीमारी की उत्पत्ति से लेकर विषमयुग्मक अप्रभावी व्यक्तियों की आवृत्ति तक पीछे जाने के लिये हार्डी-वीनबर्ग सिद्धांत का प्रयोग कर सकते हैं।
दायरा और सैद्धांतिक विचार[संपादित करें]
जनसंख्या आनुवांशिकी के गणित का विकास मूलतः आधुनिक विकासमूलक संश्लेषण के एक भाग के रूप में हुआ था। बेट्टी (Beatty) (1986) के अनुसार, यह आधुनिक संश्लेषण के मूल को परिभाषित करता है।
लेवोंटिन (Lewontin) (1974) के अनुसार, जनसंख्या आनुवांशिकी का सैद्धांतिक कार्य दो अंतरालों की एक प्रक्रिया है: एक "जेनोटाइपिक अंतराल (genotypic space)" और एक "फेनोटाइपिक अंतराल (phenotypic space)". जनसंख्या आनुवांशिकी के एक पूर्ण सिद्धांत की चुनौती नियमों का एक ऐसा समुच्चय प्रदान करना है, जो जेनोटाइप (G 1) वाली एक जनसंख्या का मिलान फेनोटाइप (P 1), जहां चयन किया जाता है, के साथ पूर्वानुमेय रूप से कर सके और नियमों एक अन्य समुच्चय, जो परिणामित जनसंख्या (P 2) का मिलान पुनः जेनोटाइप (G 2) के साथ कर सके, जहां मेंडेलीय आनुवांशिकी के द्वारा जेनोटाइपों की अगली पीढ़ी का पूर्वानुमान लगाया जा सकता है और इस तरह चक्र पूरा हो जाता है। एक क्षण के लिये यदि हम आण्विक आनुवांशिकी के गैर-मेंडेलीय (non-Mendelian) पहलुओं को एक ओर रख दें, तो भी स्पष्ट रूप से यह एक विशाल कार्य है। इस रूपांतरण को योजनाबद्ध रूप से देखने पर:
(लेवोंटिन (Lewontin) 1974, पृष्ठ+ 12 से अनुकूलित). एक्सडी (XD)
T 1 आनुवांशिक (genetic) और एपिजेनेटिक (epigenetic) नियमों, कार्यात्मक जीवविज्ञान के पहलुओं, या विकास का प्रतिनिधित्व करता है, जो एक जेनोटाइप को फेनोटाइप में रूपांतरित करते हैं। हम इसका उल्लेख "जेनोटाइप-फेनोटाइप मानचित्र" के रूप में करेंगे। T 2 प्राकृतिक चयन के कारण होने वाला रूपांतरण है, T 3 एपिजेनेटिक संबंध हैं, जो चयनित फेनोटाइप के आधार पर जेनोटाइप का पूर्वानुमान लगाते हैं और अंततः T 4 मेंडेलीय आनुवांशिकी के नियम हैं।
व्यवहार में, विकासमूलक सिद्धांत के दो भाग हैं, जो एक दूसरे के समानांतर हैं, जेनोटाइप अंतराल में कार्यरत पारंपरिक जनसंख्या आनुवांशिकी और वनस्पतियों तथा पशुओं के प्रजनन में प्रयुक्त बायोमेट्रिक सिद्धांत, जो फेनोटाइप अंतराल में कार्यरत है। यहां जेनोटाइप और फेनोटाइप अंतराल के बीच का मानचित्रण अनुपस्थित है। इसका कारण "हाथ की सफाई" है (जैसा की लेवोंटिन ने इसका ज़िक्र किया है), जिसके द्वारा एक क्षेत्र की परिवर्तनीय वस्तुओं (variables) को मापदंड या स्थिर वस्तुएं (constants) माना जाता है, जहां, एक पूर्ण-व्यवहार में, वे विकासमूलक प्रक्रिया के द्वारा स्वतः ही रूपांतरित हो जाएंगे और वास्तविकता में वे किसी अन्य क्षेत्र की अवस्था को परिवर्तित करनेवाले कार्य (functions) हैं। इस "हाथ की सफाई" में यह माना जाता है कि हम इस मानचित्रण को जानते हैं। ऐसा मान लेना कि हम इसे समझ रहे हैं, हमारी रुचि के अनेक मामलों का विश्लेषण करने के लिये पर्याप्त है। उदाहरण के लिये, यदि फेनोटाइप जेनोटाइप के साथ लगभग एक-प्रति-एक (one-to-one) (सिकल-सेल रोग) हो या समय-माप पर्याप्त रूप से संक्षिप्त हो, तो "स्थिर वस्तुओं" को उसी रूप में स्वीकार किया जा सकता है, जिस रूप में वे मौजूद हों; हालांकि, अनेक स्थितियों में ऐसा करना गलत भी होता है।
चार प्रक्रियाएं[संपादित करें]
प्राकृतिक चयन[संपादित करें]
प्राकृतिक चयन (Natural selection) वह प्रक्रिया है, जिसके द्वारा किसी जीव के अस्तित्व और सफल प्रजनन की संभावना को बढ़ाने वाले वंशानुक्रमिक लक्षण किसी जनसंख्या की आने वाली पीढ़ियों में अधिक आम बन जाते हैं।
जीवों की एक जनसंख्या के भीतर प्राकृतिक आनुवांशिक विविधता का अर्थ यह है कि वर्तमान वातावरण में कुछ जीव अन्य जीवों की तुलना में अधिक सफलतापूर्वक जीवित बचे रहेंगे यह मुद्दा चार्ल्स डार्विन ने लैंगिक चयन के अपने विचारों में विकसित किया कि प्रजननात्मक सफलता को प्रभावित करने वाले कारक भी महत्वपूर्ण हैं।
प्राकृतिक चयन फेनोटाइप पर, या जीव के देखे जा सकने वाले लक्षणों पर कार्य करता है, लेकिन किसी भी फेनोटाइप का आनुवांशिक (पैतृक) आधार, जो कि प्रजनन का लाभ प्रदान करता है, एक जनसंख्या में अधिक आम बन जाएगा (युग्म-विकल्पी आवृत्ति देखें). समय के साथ-साथ, इस प्रक्रिया का परिणाम किसी विशिष्ट पारिस्थितिक आवास के अनुकूलनों के रूप में मिल सकता है और अंततः इसके परिणामस्वरूप एक नई प्रजाति का जन्म भी हो सकता है।
प्राकृतिक चयन आधुनिक जीव-विज्ञान की आधारशिलाओं में से एक है। यह नई शब्दावली चार्ल्स डार्विन ने 1859 की अपनी अभूतपूर्व पुस्तक ऑन द ओरिजिन ऑफ स्पीशीज़ (On the Origin of Species) में प्रस्तुत की थी,[4] जिसमें प्राकृतिक चयन का वर्णन कृत्रिम चयन, एक प्रक्रिया जिसके अंतर्गत पशुओं व वनस्पतियों को उनके मनुष्य प्रजनकों द्वारा वांछित लक्षणों के द्वारा प्रजनन के लिये व्यवस्थित ढंग से समर्थन दिया जाता है, के साथ इसकी तुलना के द्वारा किया गया था। मूलतः प्राकृतिक चयन की अवधारणा का विकास आनुवांशिकता के एक मान्य सिद्धांत के अभाव में हुआ था; डार्विन के लेखन के समय, आधुनिक आनुवांशिकी विज्ञान के बारे में कोई जानकारी नहीं थी। डार्विनियाई विकास तथा इसके बाद पारंपरिक और आण्विक आनुवांशिकी में हुई खोजों के आपसी संयोजन को ही आधुनिक विकासात्मक संश्लेषण (modern evolutionary synthesis) कहा जाता है। प्राकृतिक चयन अनुकूलनात्मक विकास का मुख्य कारण बना हुआ है।
आनुवांशिक झुकाव[संपादित करें]
आनुवांशिक झुकाव (Genetic drift) उस सापेक्ष आवृत्ति में होने वाला अंतर है, जिसमें किसी जीन का एक अन्य रूप (युग्म-विकल्पी) यादृच्छिक सैंपलिंग और अवसर के कारण किसी जनसंख्या में उत्पन्न होता है। अर्थात, जनसंख्या में आने वाली पीढ़ियों में उपस्थित युग्म-विकल्पी इनके अभिभावकों में उपस्थित युग्म-विकल्पियों के एक यादृच्छिक सैंपल होते हैं। और इस बात के निर्धारण में अवसर (chance) की भूमिका होती है कि क्या किसी विशिष्ट जीव का अस्तित्व बचा रहेगा और वह प्रजनन करेगा। एक जनसंख्या की युग्म-विकल्पी आवृत्ति किसी विशिष्ट रूप को साझा करने वाले जीन युग्म-विकल्पियों की कुल संख्या की तुलना में इसकी जीन प्रतियों का भाग या प्रतिशत होता है।[5]
आनुवांशिक झुकाव एक महत्वपूर्ण विकासात्मक प्रक्रिया है, जिसके परिणामस्वरूप समय के साथ-साथ युग्म-विकल्पी आवृत्तियों में परिवर्तन होते हैं। इसके कारण जीन की कोई विविधता पूर्णतः समाप्त भी हो सकती है, जिससे आनुवांशिक परिवर्तनशीलता में कमी आती है। प्राकृतिक चयन, जो जीन के विविध रूपों को प्रजनन में उनकी सफलता के आधार पर अधिक आम या कम आम बनाता है,[6] के विपरीत आनुवांशिक झुकाव के कारण होने वाले परिवर्तन वातावरण के या अनुकूलन के दबावों द्वारा संचालित नहीं होते और वे प्रजनन की सफलता के प्रति लाभदायक, तटस्थ या हानिकारक भी हो सकते हैं।
आनुवांशिक झुकाव का प्रभाव छोटी जनसंख्या में बड़ा और बड़ी जनसंख्या में छोटा होता है। प्राकृतिक चयन की तुलना में आनुवांशिक झुकाव के महत्व को लेकर वैज्ञानिकों के बीच जोरदार बहस जारी है। रोनाल्ड फिशर (Ronald Fisher) का दृष्टिकोण यह है कि आनुवांशिक झुकाव विकास में एक नगण्य भूमिका निभाता है और यही दृष्टिकोण कई दशकों तक प्रभावी रहा है। सन 1968 में मोटू किमुरा (Motoo Kimura) ने आण्विक विकास के तटस्थ सिद्धांत, जिसमें दावा किया गया है कि आनुवांशिक सामग्री में होने वाले अधिकांश परिवर्तन आनुवांशिक झुकाव के कारण होते हैं, के द्वारा इस बहस को पुनः शुरु कर दिया। [7]
उत्परिवर्तन[संपादित करें]
उत्परिवर्तन एक कोशिका के जीनोम के डीएनए (DNA) क्रम में होने वाले परिवर्तन हैं और ये विकिरण, विषाणुओं, ट्रांस्पोसोन और उत्परिवर्तक रसायनों तथा साथ ही मीयोसिस (meiosis) या डीएनए (DNA) प्रतिलिपिकरण के दौरान उत्पन्न होने वाली त्रुटियों के कारण होते हैं।[8][9][10] त्रुटियां अक्सर डीएनए (DNA) प्रतिलिपिकरण की प्रक्रिया के दौरान, दूसरे रेशे के बहुलकीकरण (polymerization) में, उत्पन्न होती हैं। ये त्रुटियां स्वयं जीव द्वारा भी अति-उत्परिवर्तन (hypermutation) जैसी कोशिकीय प्रक्रियाओं द्वारा उत्पन्न की जा सकतीं हैं।
उत्परिवर्तन किसी जीव के फेनोटाइप पर प्रभाव डाल सकते हैं, विशेष रूप से यदि वे किसी जीन के प्रोटीन कोडिंग क्रम के भीतर उत्पन्न होते हैं। डीएनए (DNA) पोलीमरेज़ की “प्रूफरीडिंग” क्षमता के कारण त्रुटियों की दर सामान्यतः बहुत कम (प्रत्येक 10 मिलियन-100 मिलियन के आधारों में 1 त्रुटि) होती है।[11][12] प्रूफरीडिंग के बिना, त्रुटियों की दरें इससे हज़ार गुना अधिक होती हैं। डीएनए (DNA) में होने वाली रासायनिक क्षति प्राकृतिक रूप से भी होती है और कोशिकाएं डीएनए (DNA) में इस टूटन और गलत मिलानों को सुधारने के लिये डीएनए (DNA) सुधार कार्यविधि का प्रयोग करती है। इसके बावजूद, कभी-कभी यह सुधार डीएनए (DNA) को इसके मूल क्रम में वापस लाने में विफल रहता है।
डीएनए (DNA) की अदला-बदली करने और जीनों को पुनः संयोजित करने के लिये गुणसूत्रीय बदलाव का प्रयोग करने वाले जीवों में, मीयोसिस के दौरान संरेखण में होने वाली त्रुटियों के कारण भी उत्परिवर्तन हो सकते हैं।[13] बदलाव में होने वाली त्रुटियां उत्पन्न होने की संभावना तब होती है, जब एक जैसे क्रम अपने सहभागी गुण-सूत्रों को गलत संरेखन अपनाने पर बाध्य करते हैं; इसके कारण जीनोम के कुछ क्षेत्र इस प्रकार होने वाले उत्परिवर्तन के प्रति अधिक संवेदनशील बन जाते हैं। ये त्रुटियां डीएनए (DNA) क्रम में बड़े संरचनात्मक बदलाव उत्पन्न करती हैं—संपूर्ण क्षेत्र का दोहराव, उत्क्रमण या विलोपन, अथवा विभिन्न गुण-सूत्रों के बीच पूरे भागों की गलती से अदला-बदली हो जाना (जिसे ट्रांसलोकेशन कहा जाता है).
उत्परिवर्तन के कारण डीएनए (DNA) क्रम में विभिन्न प्रकार के अनेक परिवर्तन हो सकते हैं; संभव है कि इनका कोई प्रभाव न हो, या ये एक जीन के उत्पाद को परिवर्तित कर दें या ये जीन को कार्य करने से रोक दें। ड्रोसोफिला मेलैनोगास्टर (Drosophila melanogaster) नामक मक्खी पर किये गये अध्ययन में यह ज्ञात हुआ कि यदि कोई उत्परिवर्तन एक जीन द्वारा उत्पन्न किसी प्रोटीन को परिवर्तित करता है, तो संभवतः यह हानिकारक होगा क्योंकि इस उत्परिवर्तन के लगभग 70 प्रतिशत भाग का प्रभाव हानिकारक होगा और शेष भाग या तो तटस्थ होगा अथवा बहुत ही कम लाभदायक होगा। [14] कोशिकाओं पर इन उत्परिवर्तनों के संभावित हानिकारक प्रभावों के कारण, जीवों ने उत्परिवर्तनों को दूर करने के लिये डीएनए (DNA) सुधार जैसी कार्यविधियां विकसित कर लीं हैं।[8] अतः प्रजातियों के बीच उत्परिवर्तन की इष्टतम दर उच्च उत्परिवर्तन दर, जैसे क्षतिकर उत्परिवर्तन, की लागतों और उत्परिवर्तन दर को कम करने के लिये प्रणालियों, जैसे डीएनए (DNA) सुधार किण्वकों, के रख-रखाव की चयापचयी लागतों के बीच होती है।[15] अपने आनुवांशिक पदार्थ के रूप में आरएनए (RNA) का प्रयोग करने वाले विषाणुओं में उत्परिवर्तन की दरें तीव्र होती हैं,[16] जिससे लाभ हो सकता है क्योंकि ये विषाणु लगातार तथा तीव्र गति से विकसित होंगे और इस प्रकार, उदाहरणार्थ मानवीय प्रतिरक्षा तंत्र की, रक्षात्मक प्रतिक्रिया से बच निकलेंगे.[17]
उत्परिवर्तन में डीएनए (DNA) के बड़े भागों का दोहराव शामिल हो सकता है, जो कि सामान्यतः आनुवांशिक पुनर्संयोजन के माध्यम से होता है।[18] ये दोहराव नये जीनों के निर्माण के लिये कच्चे माल का एक मुख्य स्रोत हैं और प्रति दस लाख वर्षों में पशु जीनोम में दसियों से सैकड़ों जीनों को प्रतिलिपित किया जाता है।[19] अधिकांश जीन साझा वंश के जीनों के बड़े परिवारों के सदस्य होते हैं।[20] नये जीनों का उत्पादन विभिन्न विधियों के द्वारा किया जाता है, सामान्यतः एक पैतृक जीन के दोहराव और उत्परिवर्तन के माध्यम से, अथवा विभिन्न जीनों के भागों को पुनर्संयोजित करके नये कार्य करने वाले नये संयोजनों के निर्माण के द्वारा.[21][22]
यहां, क्षेत्र खण्डों के रूप में कार्य करते हैं, जिनमें से प्रत्येक का एक विशिष्ट और स्वतंत्र कार्य होता है और जिन्हें आपस में मिलाकर नये गुणों के साथ नये प्रोटीन को कूटबद्ध करने वाले जीन उत्पन्न किये जा सकते हैं।[23] उदाहरण के लिये, मनुष्य की आंख प्रकाश को पहचान पाने वाली संरचना का निर्माण करने के लिये चार जीनों का प्रयोग करती है: तीन रंगीन दृष्टि के लिये और एक रात्रिकालीन दृष्टि के लिये; ये चारों एक ही पूर्वज जीन से उत्पन्न होते हैं।[24] एक जीन (या यहां तक कि पूरे जीनोम) की प्रतिलिपि बनाने का एक अन्य लाभ यह है कि इससे अतिरेक बढ़ता है; यह युग्म के एक जीन को नये कार्य को अपनाने की अनुमति देता है, जबकि अन्य प्रतिलिपि मूल कार्य जारी रखती है।[25][26] उत्परिवर्तन के अन्य प्रकार अक्सर पूर्व में कोडिंग न किये गये डीएनए (DNA) से नये जीन का निर्माण करते हैं।[27][28]
जीन प्रवाह[संपादित करें]
जीन प्रवाह जनसंख्याओं, जो कि अक्सर समान प्रजातियां होतीं हैं, के बीच जीनों की अदला-बदली है।[29] एक समान प्रजातियों के बीच जीन प्रवाह के उदाहरणों में जीवों का प्रवसन और इसके बाद प्रजनन, अथवा पराग-कणों की अदला-बदली शामिल हैं। प्रजातियों के बीच जीन स्थानांतरण में संकर जीवों का निर्माण और क्षैतिज जीन स्थानांतरण शामिल हैं।
जनसंख्या के भीतर या बाहर प्रवसन युग्म-विकल्पियों की आवृत्ति को बदल सकता है और साथ ही जनसंख्या में नई आनुवांशिक विविधतायें भी प्रस्तुत कर सकता है। आप्रवासन किसी जनसंख्या के जीन पूल में नये आनुवांशिक पदार्थ जोड़ सकता है। इसके विपरीत उत्प्रवास आनुवांशिक पदार्थ को हटा सकता है। चूंकि जनसंख्याओं की नई प्रजातियों के निर्माण के लिये दो छितरी हुई जनसंख्याओं के बीच प्रजनन के प्रति अवरोधों की आवश्यकता होती है, जीन प्रवाह जनसंख्याओं के बीच आनुवांशिक अंतर फैलाकर इस प्रक्रिया को धीमा कर सकता है। पर्वत-श्रृंखलाओं, महासागरों और रेगिस्तानों या यहां तक कि मानव-निर्मित संरचनाओं, जैसे चीन की विशाल दीवार, जिसने वनस्पति के जीनों के प्रवाह को रोक दिया, जीन प्रवाह में अवरोध उत्पन्न करते हैं।[30]
इस आधार पर कि दो प्रजातियां अपने सबसे हालिया आम पूर्वज से कितनी दूर हट गईं हैं, अपनी संतान उत्पन्न करना उनके लिये अभी भी संभव हो सकता है, जैसे घोड़ों और गधों के बीच प्रजनन से खच्चर उत्पन्न होते हैं।[31] आम तौर पर ऐसे संकर जीव अनुपजाऊ होते हैं क्योंकि गुण-सूत्रों के दो भिन्न समुच्चय मीयोसिस के दौरान जोड़ियां बना पाने में सक्षम नहीं होते. इस स्थिति में, निकट संबद्ध प्रजातियां नियमित रूप से संकरण कर सकतीं हैं, लेकिन संकर जीव इसके विपरीत चुने जाएंगे और प्रजातियां विशिष्ट बनीं रहेंगी. हालांकि, कभी-कभी वर्धनक्षम संकर जीव भी उत्पन्न होते हैं और इन नई प्रजातियों में अपनी अभिभावक प्रजातियों के मध्यवर्ती गुण होते हैं, अथवा उनमें पूर्णतः नए फेनिटाइप होते हैं।[32] पशुओं की नई प्रजातियों के निर्माण में संकरण (hybridization) का महत्व अस्पष्ट है, हालांकि, इसके मामले अनेक प्रकार के पशुओं में देखे जाते रहे हैं,[33] और ग्रे ट्री मेंढक इसका एक विशिष्ट उदाहरण है, जिसका बहुत अच्छी तरह अध्ययन किया गया है।[34]
तथापि संकरण वनस्पतियों में प्रजातिकरण का एक महत्वपूर्ण माध्यम है, क्योंकि पशुओं की तुलना में वनस्पतियों द्वारा पॉलीप्लॉइडी (प्रत्येक गुणसूत्र की दो से अधिक प्रतियां होना) को अधिक स्वीकार किया जाता है।[35][36] पॉलीप्लॉइडी संकरणों में महत्वपूर्ण है क्योंकि यह ऐसे प्रजनन की अनुमति देता है, जिसमें मीयोसिस के दौरान गुणसूत्रों के दो भिन्न समुच्चयों में से प्रत्येक एक समरूप सहभागी के साथ जोड़ी बनाने में सक्षम होता है।[37] पॉलीप्लॉइड जीवों में अधिक आनुवांशिक विविधता भी होती है, जिसके कारण वे छोटी जनसंख्याओं में अंतः प्रजनन के दबाव से बचे रह सकते हैं।[38]
क्षैतिज जीन स्थानांतरण एक जीव से किसी दूसरे ऐसे जीव में आनुवांशिक सामग्री का स्थानांतरण है, जो उसकी संतान न हो; यह जीवाणुओं में सबसे आम है।[39] चिकित्सा के क्षेत्र में, यह एंटीबायोटिक के प्रति प्रतिरोध के विस्तार में योगदान करता हैम क्योंकि जब एक जीवाणु प्रतिरोधी जीन प्राप्त कर लेता है, तो वह तेज़ी से उन्हें दूसरी प्रजातियों तक स्थानांतरित कर सकता है।[40] जीवाणुओं से यूकेरियोट (eukaryote) जीवों, जैसे खमीर सैकहेरोमाइसेस सेरेविसी (Saccharomyces cerevisiae) और आज़ुकी बीन भंवरे कैलोसोब्रुकस चाइनेन्सिस (Callosobruchus chinensis) तक जीनों का क्षैतिज स्थानांतरण भी हुआ होगा। [41][42] बड़े पैमाने पर हुए स्थानांतरणों का एक उदाहरण यूकेरियॉटिक डेलॉइड रॉटिफर (bdelloid rotifers) हैं, जिन्होंने शायद जीवाणुओं, कवकों तथा वनस्पतियों से जीनों की एक श्रृंखला प्राप्त की है।[43] विषाणु भी जीवों के बीच डीएनए (DNA) ले जा सकते हैं, जिससे जैविक क्षेत्रों के बीच भी जीनों का स्थानांतरण होता है।[44] क्लोरोप्लास्ट व माइटोकॉन्ड्रिया के अधिग्रहण के दौरान यूकेरियोटिक (eukaryotic) कोशिकाओं तथा प्रोकेरियोट जीवों (prokaryotes) के बीच भी बड़े पैमाने पर जीन स्थानांतरण हो सकता है।[45]
एक जनसंख्या से दूसरी में युग्म-विकल्पियों के स्थानांतरण को जीन प्रवाह कहते हैं।
किसी एक जनसंख्या के भीतर या उससे बाहर प्रवासन युग्म-विकल्पी आवृत्तियों में एक लक्षणीय परिवर्तन के लिये उत्तरदायी हो सकता है। आप्रवासन के परिणामस्वरूप भी किसी विशिष्ट प्रजाति या जनसंख्या के स्थापित जीन पूल में नये आनुवांशिक भिन्न-रूप (variants) जुड़ सकते हैं।
विभिन्न जनसंख्याओं के बीच जीन प्रवाह की दर को प्रभावित करने वाले अनेक कारक हैं। सर्वाधिक महत्वपूर्ण कारकों में से एक है गतिशीलता, क्योंकि जीव की गतिशीलता जितनी अधिक होगी, उसकी प्रवासन संभावना भी उतनी ही अधिक होगी। पशुओं की गतिशीलता वनस्पतियों से अधिक होती है, हालांकि पराग-कण और बीच पशुओं या हवा के द्वारा बहुत लंबी दूरियों तक ले जाये जा सकते हैं।
दो जनसंख्याओं के बीच अनुरक्षित जीन प्रवाह के कारण भी दो जीन पूलों का संयोजन हो सकता है, जिससे दो समूहों के बीच आनुवांशिक विविधता में कमी आती है। यही कारण है कि इन समूहों के जीन पूलों का पुनर्संयोजन करके और इस प्रकार आनुवांशिक विविधता, जिसके परिणामस्वरूप पूर्ण प्रजातिकरण तथा मादा प्रजातियों का निर्माण हुआ होता, में अंतर विकसित करके जीन प्रवाह प्रजातिकरण के खिलाफ दृढ़ता से कार्य करता है।
उदाहरण के लिये, यदि घास की एक प्रजाति किसी सड़क-मार्ग के दोनों ओर विकसित हो जाती है, तो इस बात का संभावना है कि पराग-कण एक से दूसरी ओर या इसकी विपरीत दिशा में स्थानांतरित होंगे। यदि यह पराग-कण अपने गंतव्य पर प्राप्त होने वाले पौधे को निषेचित करने तथा एक वर्धनक्षम संतान उत्पन्न कर पाने में सक्षम है, तो उस पराग-कण के युग्म-विकल्पी सड़क के एक ओर की जनसंख्या से दूसरी ओर जा पाने में प्रभावी रूप से सक्षम साबित हुए हैं।
आनुवांशिक संरचना[संपादित करें]
प्रवासन में आने वाली भौतिक बाधाओं, तथा साथ ही सीमित चलायमान क्षमता, एवं पैदाइशी फाइलोपैट्री (natal philopatry) के कारण, प्राकृतिक जनसंख्याओं का पैनमिक्टिक (panmictic) होना दुर्लभ होता है (बस्टन व अन्य, 2007). सामान्यतः एक भौगोलिक सीमा होती है, जिसके भीतर रहने वाले जीव किसी सामान्य जनसंख्या से यादृच्छिक रूप से चुने गये जीवों की तुलना में एक-दूसरे से अधिक निकटता से संबंधित होते हैं। इसे उस सीमा के रूप में वर्णित किया जाता है, जहां तक कोई जनसंख्या आनुवांशिक रूप से संरचित है (रिपैकी व अन्य, 2007).
सूक्ष्मजीव जनसंख्या आनुवांशिकी[संपादित करें]
सूक्ष्मजीव जनसंख्या आनुवांशिकी शोध का एक अन्य तेज़ी से बढ़ता क्षेत्र है, जिसकी प्रासंगिकता वैज्ञानिक शोधों के अनेक अन्य सैद्धांतिक व प्रायोगिक क्षेत्रों में है। सूक्ष्मजीवों की जनसंख्या आनुवांशिकी एंटीबायोटिक प्रतिरोध तथा अत्यधिक संक्रामक रोगाणुओं के विकास का निरीक्षण करने के लिये आधार का निर्माण करती है। सूक्ष्मजीवों की जनसंख्या आनुवांशिकी लाभकारी सूक्ष्मजीवों के संरक्षण और बेहतर उपयोग के लिये रणनीतियां बनाने के लिये भी एक आवश्यक कारक है (ज़ू (Xu), 2010).
इतिहास[संपादित करें]
जनसंख्या आनुवांशिकी[संपादित करें]
जनसंख्या आनुवांशिकी का विकास मेंडेलियाई और बायोमैट्रिशियन मॉडलों के बीच एक सामंजस्य के रूप में हुआ था। ब्रिटिश जीव-विज्ञानी और सांख्यिकीविद् आर. ए. फिशर (R.A. Fisher) का कार्य इसका एक महत्वपूर्ण कदम था। अपने शोध-पत्रों की एक श्रृंखला, जिसकी शुरुआत 1918 में हुई व समाप्ति 1930 की उनकी पुस्तक द जेनेटिकल थ्योरी ऑफ नैचुरल सलेक्शन (The Genetical Theory of Natural Selection) के साथ हुई, में फिशर ने दर्शाया कि बायोमेट्रिशियनों द्वारा लगातार मापे जाने वाले अंतर अनेक असतत जीनों के संयोजित कार्य के कारण उत्पन्न हो सकते हैं और प्राकृतिक चयन किसी जनसंख्या में जीन आवृत्तियों को परिवर्तित कर सकता है, जिसके परिणामस्वरूप विकास होता है (हालांकि उस दौर में जीन के वास्तविक स्वरूप के बारे में ज्ञान की कमी के कारण, ऐसा कहा जाना चहिये कि इस तरह उन्होंने फेनोटाइपिक झुकाव की आवृत्ति को समझा, न कि विशिष्ट रूप से पहचानी जा सकने वाली जीन आवृत्ति को). सन 1924 में शुरु हुई शोध-पत्रों की एक श्रृंखला में, एक अन्य ब्रिटिश आनुवांशिकी विज्ञानी जे. बी. एस. हाल्डेन (J.B.S. Haldane) ने सांख्यिकीय विश्लेषण को प्राकृतिक चयन के वास्तविक विश्व के उदाहरणों, जैसे काले पतंगों में औद्योगिक मेलेनिन का जमाव, पर लागू किया और दर्शाया कि प्राकृतिक चयन उससे भी अधिक तेज़ दर पर कार्य करता है, जितना कि फिशर ने अनुमान लगाया था।[46][47]
अमरीकी जीव-विज्ञानी सीवॉल राइट (Sewall Wright), जिनकी पृष्ठभूमि पशुओं के प्रजनन संबंधी प्रयोगों की थी, ने अंतः क्रिया करनेवाले जीनों के संयोजनों और आनुवांशिक झुकाव को प्रदर्शित करने वाली छोटी, अपेक्षाकृत पृथक जनसंख्याओं में अंतः प्रजनन पर ध्यान केंद्रित किया। सन 1932 में, राइट ने एक अनुकूलनात्मक भूदृश्य की अवधारणा प्रस्तुत की और तर्क दिया कि आनुवांशिक झुकाव और अंतः प्रजनन एक छोटी, पृथक की गई उप-जनसंख्या को एक अनुकूलन-योग्य उच्च-बिंदु से दूर कर सकता है, जिससे प्राकृतिक चयन को इसे विभिन्न अनुकूलन-योग्य उच्च-बिंदुओं की ओर ले जाने का अवसर मिलता है। फिशर और राइट के बीच कुछ बुनियादी मुद्दों पर असहमति थी और चयन तथा झुकाव की सापेक्ष भूमिका के बारे में अमरीकियों व ब्रिटिशों के बीच एक विवाद इस सदी के अधिकांश भाग में जारी रहा। फ्रांसीसी नागरिक गुस्ताव मेलकोट (Gustave Malécot) भी इस क्षेत्र के प्रारंभिक विकास में महत्वपूर्ण थे।
फिशर, हाल्डेन और राइट के कार्य ने जनसंख्या आनुवांशिकी के क्षेत्र की बुनियाद रखी. इसने प्राकृतिक चयन को मेंडेलियाई आनुवांशिकी के साथ एकीकृत कर दिया, जो इस बात के एक एकीकृत सिद्धांत के विकास में पहला महत्वपूर्ण चरण था कि विकास किस प्रकार कार्य करता है।[46][47]
जॉन मेनार्ड स्मिथ (John Maynard Smith) हाल्डेन के विद्यार्थी थे, जबकि डब्ल्यू. डी. हैमिल्टन (W.D. Hamilton) पर फिशर के लेखन का बहुत अधिक प्रभाव था। अमरीकी नागरिक जॉर्ज आर. प्राइस (George R. Price) ने हैमिल्टन और मेनार्ड स्मिथ दोनों के साथ कार्य किया। अमरीकी रिचर्ड लेवोंटिन (Richard Lewontin) तथा जापान के मोटू किमुरा (Motoo Kimura) राइट से बहुत अधिक प्रभावित थे।
आधुनिक विकासमूलक संश्लेषण[संपादित करें]
बीसवीं सदी के कुछ शुरुआती दशकों में, अधिकांश क्षेत्र प्रकृति-विज्ञानी यह मानते रहे कि विकास की लैमार्कियाई (Lamarckian) और ऑर्थोगोनिक (orthogenic) कार्यविधियां जीवित विश्व में दिखाई देने वाली जटिलता की सर्वश्रेष्ठ व्याख्या प्रस्तुत करतीं थीं। हालांकि, जैसे-जैसे आनुवांशिकी के क्षेत्र का विकास होता गया, ये दृष्टिकोण कम मान्य बनते गये।[48] थियोडॉसियस डोब्ज़ैंस्की (Theodosius Dobzhansky), टी. एच. मॉर्गन (T. H. Morgan) की प्रयोगशाला में एक शोध-कर्ता, आनुवांशिक विविधता के क्षेत्र में रूसी आनुवांशिकी विज्ञानियों, जैसे सर्गेई चेत्वेरिकोव (Sergei Chetverikov), द्वारा किये गये कार्यों से प्रभावित रहे थे। 1937 की अपनी पुस्तक जेनेटिक्स एंड ओरिजिन ऑफ स्पीशीज़ (Genetics and the Origin of Species) के द्वारा उन्होंने जनसंख्या आनुवांशिकी-विज्ञानियों द्वारा विकसित सूक्ष्म-विकास के आधार तथा क्षेत्र जीव-विज्ञानियों द्वारा देखे गये वृहत-विकास के पैटर्न के बीच की दूरी को पाटने में सहायता की।
डोब्ज़ैंस्की (Dobzhansky) ने जंगली जनसंख्याओं की आनुवांशिक विविधता का परीक्षण किया और यह दर्शाया कि, जनसंख्या आनुवांशिकी की धारणाओं के विपरीत, इन जनसंख्याओं में बड़ी मात्रा में आनुवांशिक विविधता थी और साथ ही उप-जनसंख्याओं के बीच भी अंतर देखे जा सकते थे। इस पुस्तक में जनसंख्या आनुवांशिकी का अत्यधिक गणितीय कार्य भी शामिल था और इसे एक अधिक अभिगम्य रूप में रखा गया था। ग्रेट ब्रिटेन में ई. बी. फोर्ड. (E.B. Ford), पारिस्थितिक आनुवांशिकी के अगुआ, ने 1930 के दशक और 1940 के दशक में पारिस्थितिक कारकों, जिनमें आनुवांशिक बहुरूपता, जैसे मनुष्य के रक्त प्रकार, के माध्यम से आनुवांशिक विविधता को बनाये रखने की क्षमता भी शामिल है, के कारण चयन की शक्ति को प्रदर्शित करना जारी रखा। फोर्ड के कार्य ने आधुनिक संश्लेषण के विकास के दौरान आनुवांशिक झुकाव की तुलना में प्राकृतिक चयन को अधिक महत्व प्रदान करने में योगदान दिया। [46][49][50]
| यह सम्पूर्ण पृष्ठ या इसके कुछ अनुभाग हिन्दी के अतिरिक्त अन्य भाषा(ओं) में भी लिखे गए हैं। आप इनका करके विकिपीडिया की सहायता कर सकते हैं। |
इन्हें भी देखें
- समाचयी थ्योरी
- ड्यूअल इन्हेरिटेंस थ्योरी
- पारिस्थितिक आनुवांशिकी
- विकासमूलक रूप से महत्वपूर्ण ईकाई
- इवेन्स का सैम्पलिंग फॉर्मुला
- फिटनेस लैंडस्केप
- संस्थापक प्रभाव
- आनुवंशिक विविधता
- आनुवंशिक झुकाव
- आनुवंशिक भूक्षरण
- आनुवंशिक हिचहाइकिंग
- आनुवंशिक प्रदूषण
- जीन पूल
- जेनोटाइप-फेनोटाइप भेद
- आवास विखंडन
- हाल्डेन का असमंजस
- हार्डी-विन्बर्ग सिद्धांत
- हिल-रॉबर्टसन प्रभाव
- श्रृंखला असंतुलन
- सूक्ष्म-विकास
- आण्विक विकास
- मुलर का रैशे
- म्युटेशनल मेल्टडाउन
- आण्विक विकास का तटस्थ सिद्धांत
- जनसंख्या मार्गावरोध
- मात्रात्मक आनुवांशिकी
- प्रजनन सम्पूर्ति
- चयन
- चयनात्मक प्रसार
- जनसंख्या का छोटा आकार
- विषाणुओं की अर्ध-प्रजातियां
सन्दर्भ[संपादित करें]
- ↑ Hartl, Daniel (2007). Principles of Population Genetics. Sinauer Associates. पृ॰ 95. आई॰ऍस॰बी॰ऍन॰ 978-0-87893-308-2.
- ↑ O'Neil, Dennis (2008). "Hardy-Weinberg Equilibrium Model". The synthetic theory of evolution: An introduction to modern evolutionary concepts and theories. Behavioral Sciences Department, Palomar College. मूल से 19 फ़रवरी 2008 को पुरालेखित. अभिगमन तिथि 2008-01-06.
- ↑ Bright, Kerry (2006). "Causes of evolution". Teach Evolution and Make It Relevant. National Science Foundation. मूल से 7 अक्तूबर 2010 को पुरालेखित. अभिगमन तिथि 2007-12-30.
- ↑ डार्विन सी (1859) ऑन द ओरिजिन ऑफ़ स्पिशिज़ बाई मीन्स ऑफ़ नैचरल सिलेक्शन और द प्रिवेंशन ऑफ़ फेवर्ड रेसेस इन द स्ट्रगल फॉर लाइफ जॉन मर्रे, लंडन; मॉडर्न रिप्रिंट Charles Darwin, Julian Huxley (2003). The Origin of Species. Signet Classics. आई॰ऍस॰बी॰ऍन॰ 0-451-52906-5. पब्लिश्ड ऑनलाइन एट द कम्प्लीट वर्क ऑफ़ चार्ल्स डार्विन ऑनलाइन Archived 2005-06-29 at the वेबैक मशीन: ऑन द ओरिजिन ऑफ़ स्पिशिज़ बाई मीन्स ऑफ़ नैचरल सिलेक्शन और द प्रिज़र्वेशन ऑफ़ फेवर्ड रेसेस इन द स्ट्रगल ऑफ़ लाइफ Archived 2011-08-13 at the वेबैक मशीन.
- ↑ Futuyma, Douglas (1998). Evolutionary Biology. Sinauer Associates. पृ॰ Glossary. आई॰ऍस॰बी॰ऍन॰ 0-87893-189-9.
- ↑ Avers, Charlotte (1989). Process and Pattern in Evolution. Oxford University Press
- ↑ Futuyma, Douglas (1998). Evolutionary Biology. Sinauer Associates. पृ॰ 320. आई॰ऍस॰बी॰ऍन॰ 0-87893-189-9.
- ↑ अ आ Bertram J (2000). "The molecular biology of cancer". Mol. Aspects Med. 21 (6): 167–223. PMID 11173079. डीओआइ:10.1016/S0098-2997(00)00007-8.
- ↑ Aminetzach YT, Macpherson JM, Petrov DA (2005). "Pesticide resistance via transposition-mediated adaptive gene truncation in Drosophila". Science. 309 (5735): 764–7. PMID 16051794. डीओआइ:10.1126/science.1112699.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Burrus V, Waldor M (2004). "Shaping bacterial genomes with integrative and conjugative elements". Res. Microbiol. 155 (5): 376–86. PMID 15207870. डीओआइ:10.1016/j.resmic.2004.01.012.
- ↑ Griffiths, William M.; Miller, Jeffrey H.; Suzuki, David T.; Lewontin, Richard C.; Gelbart, संपा॰ (2000). "Spontaneous mutations". An Introduction to Genetic Analysis (7th संस्करण). New York: W. H. Freeman. आई॰ऍस॰बी॰ऍन॰ 0-7167-3520-2.
|editor1-first=और|editor-first=के एक से अधिक मान दिए गए हैं (मदद) - ↑ Freisinger, E; Grollman, AP; Miller, H; Kisker, C (2004). "Lesion (in)tolerance reveals insights into DNA replication fidelity". The EMBO journal. 23 (7): 1494–505. PMID 15057282. डीओआइ:10.1038/sj.emboj.7600158. पी॰एम॰सी॰ 391067.
- ↑ Griffiths, William M.; Miller, Jeffrey H.; Suzuki, David T.; Lewontin, Richard C.; Gelbart, संपा॰ (2000). "Chromosome Mutation I: Changes in Chromosome Structure: Introduction". An Introduction to Genetic Analysis (7th संस्करण). New York: W. H. Freeman. आई॰ऍस॰बी॰ऍन॰ 0-7167-3520-2.
|editor1-first=और|editor-first=के एक से अधिक मान दिए गए हैं (मदद) - ↑ Sawyer SA, Parsch J, Zhang Z, Hartl DL (2007). "Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila". Proc. Natl. Acad. Sci. U.S.A. 104 (16): 6504–10. PMID 17409186. डीओआइ:10.1073/pnas.0701572104. पी॰एम॰सी॰ 1871816.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Sniegowski P, Gerrish P, Johnson T, Shaver A (2000). "The evolution of mutation rates: separating causes from consequences". Bioessays. 22 (12): 1057–66. PMID 11084621. डीओआइ:10.1002/1521-1878(200012)22:12<1057::AID-BIES3>3.0.CO;2-W.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Drake JW, Holland JJ (1999). "Mutation rates among RNA viruses". Proc. Natl. Acad. Sci. U.S.A. 96 (24): 13910–3. PMID 10570172. डीओआइ:10.1073/pnas.96.24.13910. पी॰एम॰सी॰ 24164. मूल से 13 मार्च 2013 को पुरालेखित. अभिगमन तिथि 11 अक्तूबर 2010.
- ↑ Holland J, Spindler K, Horodyski F, Grabau E, Nichol S, VandePol S (1982). "Rapid evolution of RNA genomes". Science. 215 (4540): 1577–85. PMID 7041255. डीओआइ:10.1126/science.7041255.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Hastings, P J; Lupski, JR; Rosenberg, SM; Ira, G (2009). "Mechanisms of change in gene copy number". Nature Reviews. Genetics. 10 (8): 551–564. PMID 19597530. डीओआइ:10.1038/nrg2593. पी॰एम॰सी॰ 2864001.
- ↑ Carroll SB, Grenier J, Weatherbee SD (2005). From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design. Second Edition. Oxford: Blackwell Publishing. आई॰ऍस॰बी॰ऍन॰ 1-4051-1950-0.
|author=और|last=के एक से अधिक मान दिए गए हैं (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Harrison P, Gerstein M (2002). "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution". J Mol Biol. 318 (5): 1155–74. PMID 12083509. डीओआइ:10.1016/S0022-2836(02)00109-2.
- ↑ Orengo CA, Thornton JM (2005). "Protein families and their evolution-a structural perspective". Annu. Rev. Biochem. 74: 867–900. PMID 15954844. डीओआइ:10.1146/annurev.biochem.74.082803.133029.
- ↑ Long M, Betrán E, Thornton K, Wang W (2003). "The origin of new genes: glimpses from the young and old". Nat. Rev. Genet. 4 (11): 865–75. PMID 14634634. डीओआइ:10.1038/nrg1204. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Wang M, Caetano-Anollés G (2009). "The evolutionary mechanics of domain organization in proteomes and the rise of modularity in the protein world". Structure. 17 (1): 66–78. PMID 19141283. डीओआइ:10.1016/j.str.2008.11.008.
- ↑ Bowmaker JK (1998). "Evolution of colour vision in vertebrates". Eye (London, England). 12 (Pt 3b): 541–7. PMID 9775215.
- ↑ Gregory TR, Hebert PD (1999). "The modulation of DNA content: proximate causes and ultimate consequences". Genome Res. 9 (4): 317–24. PMID 10207154. डीओआइ:10.1101/gr.9.4.317. मूल से 23 अगस्त 2014 को पुरालेखित. अभिगमन तिथि 11 अक्तूबर 2010. नामालूम प्राचल
|doi_brokendate=की उपेक्षा की गयी (|doi-broken-date=सुझावित है) (मदद) - ↑ Hurles M (2004). "Gene duplication: the genomic trade in spare parts". PLoS Biol. 2 (7): E206. PMID 15252449. डीओआइ:10.1371/journal.pbio.0020206. पी॰एम॰सी॰ 449868. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद) - ↑ Liu N, Okamura K, Tyler DM (2008). "The evolution and functional diversification of animal microRNA genes". Cell Res. 18 (10): 985–96. PMID 18711447. डीओआइ:10.1038/cr.2008.278. पी॰एम॰सी॰ 2712117. मूल से 2 फ़रवरी 2015 को पुरालेखित. अभिगमन तिथि 11 अक्तूबर 2010.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Siepel A (2009). "Darwinian alchemy: Human genes from noncoding DNA". Genome Res. 19 (10): 1693–5. PMID 19797681. डीओआइ:10.1101/gr.098376.109. पी॰एम॰सी॰ 2765273. मूल से 23 अगस्त 2014 को पुरालेखित. अभिगमन तिथि 11 अक्तूबर 2010. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद) - ↑ Morjan C, Rieseberg L (2004). "How species evolve collectively: implications of gene flow and selection for the spread of advantageous alleles". Mol. Ecol. 13 (6): 1341–56. PMID 15140081. डीओआइ:10.1111/j.1365-294X.2004.02164.x. पी॰एम॰सी॰ 2600545.
- ↑ Su H, Qu L, He K, Zhang Z, Wang J, Chen Z, Gu H (2003). "The Great Wall of China: a physical barrier to gene flow?". Heredity. 90 (3): 212–9. PMID 12634804. डीओआइ:10.1038/sj.hdy.6800237.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Short RV (1975). "The contribution of the mule to scientific thought". J. Reprod. Fertil. Suppl. (23): 359–64. PMID 1107543.
- ↑ Gross B, Rieseberg L (2005). "The ecological genetics of homoploid hybrid speciation". J. Hered. 96 (3): 241–52. PMID 15618301. डीओआइ:10.1093/jhered/esi026. पी॰एम॰सी॰ 2517139.
- ↑ Burke JM, Arnold ML (2001). "Genetics and the fitness of hybrids". Annu. Rev. Genet. 35: 31–52. PMID 11700276. डीओआइ:10.1146/annurev.genet.35.102401.085719.
- ↑ Vrijenhoek RC (2006). "Polyploid hybrids: multiple origins of a treefrog species". Curr. Biol. 16 (7): R245. PMID 16581499. डीओआइ:10.1016/j.cub.2006.03.005.
- ↑ Wendel J (2000). "Genome evolution in polyploids". Plant Mol. Biol. 42 (1): 225–49. PMID 10688139. डीओआइ:10.1023/A:1006392424384.
- ↑ Sémon M, Wolfe KH (2007). "Consequences of genome duplication". Curr Opin Genet Dev. 17 (6): 505–12. PMID 18006297. डीओआइ:10.1016/j.gde.2007.09.007.
- ↑ Comai L (2005). "The advantages and disadvantages of being polyploid". Nat. Rev. Genet. 6 (11): 836–46. PMID 16304599. डीओआइ:10.1038/nrg1711.
- ↑ Soltis P, Soltis D (2000). "The role of genetic and genomic attributes in the success of polyploids". Proc. Natl. Acad. Sci. U.S.A. 97 (13): 7051–7. PMID 10860970. डीओआइ:10.1073/pnas.97.13.7051. पी॰एम॰सी॰ 34383. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद) - ↑ Boucher Y, Douady CJ, Papke RT, Walsh DA, Boudreau ME, Nesbo CL, Case RJ, Doolittle WF (2003). "Lateral gene transfer and the origins of prokaryotic groups". Annu Rev Genet. 37: 283–328. PMID 14616063. डीओआइ:10.1146/annurev.genet.37.050503.084247.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Walsh T (2006). "Combinatorial genetic evolution of multiresistance". Curr. Opin. Microbiol. 9 (5): 476–82. PMID 16942901. डीओआइ:10.1016/j.mib.2006.08.009.
- ↑ Kondo N, Nikoh N, Ijichi N, Shimada M, Fukatsu T (2002). "Genome fragment of Wolbachia endosymbiont transferred to X chromosome of host insect". Proc. Natl. Acad. Sci. U.S.A. 99 (22): 14280–5. PMID 12386340. डीओआइ:10.1073/pnas.222228199. पी॰एम॰सी॰ 137875.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Sprague G (1991). "Genetic exchange between kingdoms". Curr. Opin. Genet. Dev. 1 (4): 530–3. PMID 1822285. डीओआइ:10.1016/S0959-437X(05)80203-5.
- ↑ Gladyshev EA, Meselson M, Arkhipova IR (2008). "Massive horizontal gene transfer in bdelloid rotifers". Science. 320 (5880): 1210–3. PMID 18511688. डीओआइ:10.1126/science.1156407. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Baldo A, McClure M (1 सितंबर 1999). "Evolution and horizontal transfer of dUTPase-encoding genes in viruses and their hosts". J. Virol. 73 (9): 7710–21. PMID 10438861. पी॰एम॰सी॰ 104298.
- ↑ Poole A, Penny D (2007). "Evaluating hypotheses for the origin of eukaryotes". Bioessays. 29 (1): 74–84. PMID 17187354. डीओआइ:10.1002/bies.20516.
- ↑ अ आ इ Bowler 2003, पृष्ठ 325–339
- ↑ अ आ Larson 2004, पृष्ठ 221–243
- ↑ Mayr & Provine 1998, पृष्ठ 295–298, 416
- ↑ Mayr, E§year=1988. Towards a new philosophy of biology: observations of an evolutionist. Harvard University Press. पृ॰ 402.
- ↑ Mayr & Provine 1998, पृष्ठ 338–341
- जे. बीटी. इंटीग्रेटिंग साइनटीफिक डिसिप्लिंस में डब्ल्यू. बिशेल और निजहॉफ डोर्डरेश द्वारा संपादित “द सिंथेसिस एंड द सिंथेटिक थ्योरी”, 1986.
- Buston, PM; एवं अन्य (2007). "Are clownfish groups composed of close relatives? An analysis of microsatellite DNA vraiation in Amphiprion percula". Molecular Ecology. 12 (3): 733–742. PMID 12675828. Explicit use of et al. in:
|author2=(मदद) - लुइगी लुका कैवेली-फोर्ज़ा. जिन्ज़, पीपल्स, एंड लैंग्वेज. नॉर्थ पॉइंट प्रेस, 2000.
- लुइगी लुका कैवेली-फोर्ज़ा एट एल. द हिस्ट्री एंड जियोग्राफी ऑफ़ ह्यूमन जिन्ज़. प्रिंसटन यूनिवर्सिटी प्रेस, 1994.
- जेम्स एफ. करो और मोटू किमोरा. जनसंख्या आनुवांशिकी सिद्धांत का परिचय. हार्पर एंड रो, 1972.
- वॉरेन जे इवेंस. गणितीय जनसंख्या आनुवांशिकी. स्प्रिंगर-वर्लग न्यूयॉर्क, इंक., 2004. ISBN 0-387-20191-2
- जॉन एच. गिलिस्पी जनसंख्या आनुवांशिकी: एक संक्षिप्त गाइड, जॉन्स हॉपकिंस प्रेस, 1998. ISBN 0-8018-5755-4.
- रिचर्ड हैलीबर्टन. जनसंख्या आनुवांशिकी का परिचय. प्रेंटिस हॉल, 2002.
- डैनियल हार्टल. जनसंख्या आनुवांशिकी के प्राइमर, तीसरा संस्करण. सिनौएर, 2000. ISBN 0-87893-304-2
- डैनियल हार्टल और एंड्रियु क्लार्क. जनसंख्या आनुवांशिकी के सिद्धांत, तीसरा संस्करण. सिनौएर, 1997. ISBN 0-87893-306-9.
- रिचर्ड सी. लेवोंटिन. विकासवादी परिवर्तन के आनुवंशिक आधार. कोलंबिया यूनिवर्सिटी प्रेस, 1974.
- विलियम बी. प्रोविन. सैद्धांतिक जनसंख्या आनुवांशिकी के मूल. शिकागो विश्वविद्यालय प्रेस. 1971. ISBN 0-226-68464-4.
- Repaci, V; Stow AJ, Briscoe DA (2007). "Fine-scale genetic structure, co-founding and multiple mating in the Australian allodapine bee (Ramphocinclus brachyurus". Journal of Zoology. 270: 687–691. डीओआइ:10.1111/j.1469-7998.2006.00191.x.
- स्पेंसर वेल्स. द जर्नी ऑफ़ मैन. रैंडम हॉउस, 2002.
- स्पेंसर वेल्स. डीप एंसेसट्री: इनसाइड द जेनोग्राफिक प्रोजेक्ट. नेशनल ज्योग्राफिक सोसाइटी, 2006.
- Cheung, KH; Osier MV, Kidd JR, Pakstis AJ, Miller PL, Kidd KK (2000). "ALFRED: an allele frequency database for diverse populations and DNA polymorphisms". Nucleic Acids Research. 28 (1): 361–3. PMID 10592274. डीओआइ:10.1093/nar/28.1.361. पी॰एम॰सी॰ 102486.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ज़ू, जे माइक्रोबियल जनसंख्या आनुवांशिकी. केस्टर अकादमिक प्रेस, 2010. ISBN 978-1-904455-59-2
बाहरी कड़ियाँ[संपादित करें]
- येल यूनिवर्सिटी में द एलेले फ्रिक्विंसी डेटाबेस
- EHSTRAFD.org – अर्थ ह्यूमन एसटीआर (STR) एलेले फ्रिक्विंसीज़ डेटाबेस
- जनसंख्या आनुवांशिकी का इतिहास
- पॉप्युलेशन के जेनेटिक कॉमपोजीशन को सिलेक्शन कैसे परिवर्तित करता है, स्टिफिंस सी. स्टियर्न्स के द्वारा लेक्चर का वीडियो (येल यूनिवर्सिटी)
- नैशनल ज्योग्राफिक: एटलस ऑफ़ द ह्यूमन जर्नी (हैप्लोग्रुप-बेस्ड ह्यूमन माइग्रेशन मैप्स)
- मोनाश वर्चुअल लैबोरेट्री – वर्चुअल लैबोरेट्री के मोनाश यूनिवर्सिटी में हैबीटैट फ्रेगमेंटेशन और पॉप्युलेशन जेनेटिक्स ऑनलाइन पर अनुकरण.



