चयापचय

उपापचय (metabolism) जीवों में जीवनयापन के लिये होने वाली रसायनिक प्रतिक्रियाओं को कहते हैं। ये प्रक्रियाएं जीवों को बढ़ने और प्रजनन करने, अपनी रचना को बनाए रखने और उनके पर्यावरण के प्रति सजग रहने में मदद करती हैं। साधारणतः उपापचय को दो प्रकारों में बांटा गया है। अपचय कार्बनिक पदार्थों का विघटन करता है, उदा. कोशिकीय श्वसन से ऊर्जा का उत्पादन. उपचय ऊर्जा का प्रयोग करके प्रोटीनों और नाभिकीय अम्लों जैसे कोशिकाओं के अंशों का निर्माण करता है।
उपापचय की रसायनिक प्रतिक्रियाएं उपापचयी मार्गों में संचालित होती हैं, जिनमें एक रसायन को एंजाइमों की श्रंखला द्वारा कुछ चरणों में दूसरे रसायन में बदला जाता है। एंजाइम उपापचय के लिये महत्वपूर्ण होते हैं, क्यौंकि वे जीवों को ऐसी अपेक्षित प्रतिक्रियाएं, जिनमें ऊर्जा की आवश्यकता होती है और जो स्वतः नहीं घट सकती हैं, उन्हें उन स्वतः होने वाली प्रतिक्रियाओं के साथ युगल रूप में होने में मदद करते हैं, जिनसे ऊर्जा उत्पन्न होती है। चूंकि एंजाइम उत्प्रेरक का काम करते हैं, इसलिये वे इन प्रतिक्रियाओं को तेजी से और य़थेष्ट रूप से होने देते हैं। एंजाइम कोशिका के पर्यावरण में परिवर्तनों या अन्य कोशिकाओं से प्राप्त संकेतों के अनुसार चयापचयी मार्गों के नियंत्रण में भी सहायता करते हैं।
किसी जीव का उपापचय यह निश्चित करता है कि उसके लिये कौन सा पदार्थ पौष्टिक होगा और कौन सा विषैला. उदा.कुछ प्रोकैर्योसाइट हाइड्रोजन सल्फाइड का प्रयोग करते हैं, जबकि यह गैस पशुओं के लिये जहरीली होती है।[1] उपापचय की गति, या उपापचय दर इस बात को भी प्रभावित करती है कि किसी जीव को कितने भोजन की जरूरत होगी.
उपापचय की एक खास बात यह है कि जातियों में बड़ी भिन्नताएं होने पर भी उनके मूल उपापचयी मार्ग और अंश समान प्रकार के होते हैं।[2] उदा. सिट्रिक एसिड चक्र में माध्यमिक भूमिका निभाने वाले कार्बाक्सिलिक एसिड, एककोशिकीय बैक्टीरिया एश्चरिशिया कोली से लेकर हाथियों जैसे विशाल बहुकोशिकीय जीवों तक, सभी में पाए जाते हैं।[3] उपापचय की ये खास समानताएं संभवतः इन मार्गों की उच्च कार्यक्षमता और विकास के इतिहास में उनके जल्दी प्रकट होने के कारण होती हैं।[4][5]
मुख्य जैवरसायन[संपादित करें]
जानवरों, पौधों और सूक्ष्मजीवों को बनाने वाली अधिकांश रचनाएं अणुओं के तीन मूल वर्गों से बनी होती हैं-अमीनो एसिड, कार्बोहाइड्रेट और लिपिड (जो वसा के नाम से भी जाना जाता है). चूंकि ये अणु जीवन के लिये महत्वपूर्ण होते हैं, इसलिये चयापचयी प्रतिक्रियाएं कोशिकाओं और ऊतकों के निर्माण के समय इन अणुओँ को बनाने, या भोजन के पाचन और प्रयोग में उन्हें विघटित करने व उन्हें ऊर्जा के स्रोत के रूप में उपयोग में लाने में जुटी होती हैं। कई महत्वपूर्ण जैवरसायन मिलकर डीएनए और प्रोटीनों जैसे पॉलिमरों का उत्पादन करते हैं। ये महाअणु अत्यावश्यक होते हैं।
| अणु का प्रकार | मोनोमर प्रकारों के नाम | पॉलिमर प्रकारों के नाम | पॉलिमर प्रकारों के उदाहरण |
|---|---|---|---|
| अमीनो एसिड | अमीनो एसिड | प्रोटीन(पॉलिपेप्टाइड) | ऱेशायुक्त प्रोटीन और ग्लॉबुलार प्रोटीन |
| कार्बोहाइड्रेट | मोनोसैक्राइड | पॉलिसैक्राइड | स्टार्च, ग्लायकोजन और सेलूलोज |
| न्यूक्लिक एसिड | न्यूक्लियोटाइड | पॉलिन्यूक्लियोटाइड | डीएनए और आरएनए |
अमीनो एसिड और प्रोटीन[संपादित करें]
प्रोटीन रैखिक श्रंखला में व्यवस्थित और पेप्टाइड बांडों द्वारा जोड़े गए अमीनो एसिडों से बने होते हैं। कई प्रोटीन चयापचय में रसायनिक प्रतिक्रियाओं को उत्प्रेरित करने वाले एंजाइम होते हैं। अन्य प्रोटीनों का कार्य रचनात्मक या प्रक्रियात्मक होता है, जैसे कोशिका पंजर बनाती है - कोशिका का आकार बनाए रखने के लिये ढांचा - बनाने वाले प्रोटीन.[6] कोशिका संकेतन, रोगनिरोधक क्षमता, कोशिकाओं के आपस में चिपकने, झिल्लियों के पार सक्रिय परिवहन और कोशिका-चक्र में भी प्रोटीनों का महत्व होता है।[7]
वसा पदार्थ[संपादित करें]
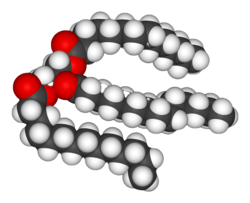
वसा पदार्थ जैवरसायनों के सबसे अधिक विविधता वाले समूह हैं। उनका मुख्य रचनात्मक उपयोग कोशिका झिल्ली जैसी जैविक झिल्लियों के भाग के रूप में, या उर्जा के स्रोत के ऱुप में होता है।[7] वसाओं को सामान्यतः हाइड्रोफोबिक या एम्फीपैथिक जैविक अणुओं के रूप में परिभाषित किया जाता है, जो बेन्ज़ीन या क्लोरोफार्म जैसे विलायकों में घुलनशील होते हैं।[8] वसा एक विशाल यौगिक समूह हैं जिनमें वसा अम्ल और ग्लिसरॉल शामिल हैं– तीन वसा अम्ल एस्टरों से जुड़े एक ग्लिसरॉल अणु को ट्यासिलग्लिसराइड कहते हैं।[9] इस मूल रचना के कई विभिन्न प्रकार पाए जाते हैं, जिनमें स्फिंगोलिपिडों में स्फिंगोसीन और हाइड्रोफिलीक समूह जैसे फास्फोलिपिडों में फास्फेट शामिल हैं। कॉलेस्ट्राल जैसे स्टीरायड, कोशिकाओं में बनने वाले वसाओं का एक और मुख्य वर्ग हैं।[10]
कार्बोहाइड्रेट[संपादित करें]
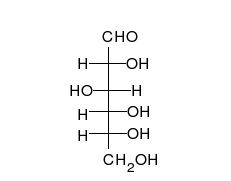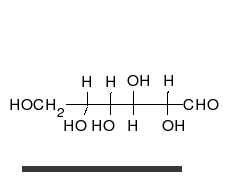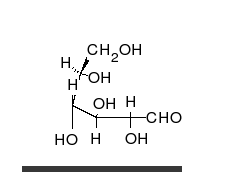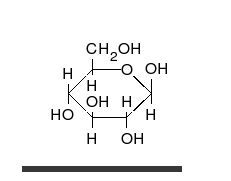
कार्बोहाइड्रेट अनेक हाइड्राक्सिल समूहों वाले सीधी श्रंखला के एल्डीहाइड या कीटोन होते हैं, जो सीधी श्रंखला या छल्लों के रूप में रह सकते हैं। कार्बोहाइड्रेट सबसे अधिक मात्रा में पाए जाने वाले जैविक अणु हैं और अनेकों भूमिकाएं निभाते हैं, जैसे ऊर्जा का संचयन और परिवहन (स्टार्च, ग्लायकोजन) और रचनात्मक भागों के रूप में (पोधों में सेलूलोज, पशुओं में काइटिन).[7] मूल कार्बोहाइड्रेट इकाइयों को मोनोसैक्राइड कहा जाता है, जिनमें गैलेक्टोज, फ्रक्टोज और सबसे महत्वपूर्ण, ग्लुकोज शामिल हैं। मोनोसैक्राइड आपस में जुड़कर लगभग असीमित रूप से पॉलिसैक्राइडों का निर्माण कर सकते हैं।[11]
न्यूक्लियोटाइड[संपादित करें]
डीएनए और आरएनए पॉलिमर न्यूक्लियोटाइडों की लंबी श्रंखलाएं होते हैं। ये अणु प्रतिलिपीकरण और प्रोटीन जैवसंश्लेषण की प्रक्रियाओं के जरिये जीन-संबंधी जानकारी के संचयन और प्रयोग के लिये आवश्यक होते हैं।[7] इस जानकारी की रक्षा डीएनए की मरम्मत प्रक्रियाओं द्वारा की जाती है और डीएनए प्रतिरूपण द्वारा संचरित की जाती है। कुछ वाइरसों जैसे एचआईवी में आरएनए जीनोम होता है, जो उल्टे प्रतिलिपीकरण का प्रयोग करके अपने वाइरल आरएनए जीनोम से डीएनए सांचे का निर्माण करता है।[12] स्प्लाइसियोसोमों और रिबोसोमों जैसे रिबोजाइमों का आरएनए एंजाइमों के समान होता है क्यौंकि यह रसायनिक प्रतिक्रियाओं को उत्प्रेरित कर सकता है। न्यूक्लियोसाइड राइबोज शुगर से नाभिकीय आधारों के जुड़ने से बनते हैं। ये आधार नाइट्रोजन युक्त हेटेरोसाइक्लिक छल्ले होते हैं, जिन्हें प्यूरीनों या पाइरिमिडीनों में वर्गीकृत किया गया है। न्यूक्लियोटाइड चयापचयी समूह अंतरण प्रतिक्रियाओं में सहएंजाइमों का काम भी करते हैं।[13]
कोएंजाइम[संपादित करें]

चयापचय में बड़ी संख्या में रसायनिक प्रतिक्रियाएं होती हैं, लेकिन उनमें से अधिकांश कार्यशील समूहों के अंतरण के लिये होने वाली चंद मूल प्रकार की प्रतिक्रियाएं होती हैं।[14] इस आम रसायनक्रिया के कारण कोशिकाएँ विभिन्न प्रतिक्रियाओं के बीच रसायनिक समूहों का वहन करने के लिये चयापचयी मध्यस्थों के छोटे से समूह का इस्तेमाल करती हैं।[13] इन समूह-अंतरण मध्यस्थों को सहएंजाइम कहा जाता है। समूह-अंतरण की प्रत्येक कक्षा एक विशेष सहएंजाइम द्वारा की जाती है, जो उसे उत्पन्न करने वाले और उसका उपयोग करने वाले एंजाइमों के सेट का सबस्ट्रेट होता है। इसलिये ये सहएंजाइम लगातार बनते, उपयोग में लिये जाते और फिर से पुनरावृत्त होते रहते हैं।[15]
एक केन्द्रीय सहएंजाइम है, एडीनोसीन ट्राईफास्फेट, जो कोशिकाओं की सर्वव्यापी ऊर्जा मुद्रा है। इस न्यूक्लियोटाइड का प्रयोग विभिन्न रसायनिक प्रतिक्रियाओं के बीच रसायनिक ऊर्जा के अंतरण के लिये किया जाता है। कोशिकाओं में एटीपी छोटी सी मात्रा में होता है, लेकिन चूंकि यह लगातार बनता रहता है, इसलिये मानव शरीर दिन भर में लगभग अपने भार के बराबर एटीपी का प्रयोग कर सकता है।[15] एटीपी अपचय और उपचय के बीच सेतु का काम करता है, जिसमें अपचय प्रतिक्रियाएं एटीपी उत्पन्न करती हैं और उपचय प्रतिक्रियाएं उसका उपयोग करती हैं। यह फास्फोरिलीकरण प्रतिक्रियाओं में फास्फेट समूहों के वाहक के रूप में भी कार्य करता है।
विटामिन छोटी मात्राओं में आवश्यक एक कार्बनिक यौगिक होता है, जो कोशिकाओं द्वारा नहीं बनाया जा सकता. मानव के पोषण में, अधिकतर विटामिन संशोधन के बाद सहएंजाइमों का कार्य करते हैं, उदा.सभी जल में घुलनशील विटामिन कोशिकाओं में प्रयोग के समय फास्फोरिलीकृत होते हैं या न्यूक्लियोटाइडों से युग्मित हो जाते हैं।[16] विटामिन बी3 (नियासिन) का एक यौगिक, निकोटिनमाइड एडीनाइन डाईन्यूक्लियोटाइड (एनएडीएच), एक महत्वपूर्ण सहएंजाइम है, जो हाइड्रोजन ग्राहक का काम करता है। सैकड़ों भिन्न प्रकार के डीहाइड्रोजनेज उनके सबस्ट्रेटों से इलेक्ट्रानों को निकाल कर NAD+ को एनएडीएच में अपघटित कर देते हैं, सहएंजाइम का यह अपघटित प्रकार कोशिकाओं के किसी भी रिडक्टेजों के लिये सबस्ट्रेट का काम करता है, जिन्हें उनके सबस्ट्रेटों का अपघटन करना होता है।[17] निकोटिनामाइड अडीनाइन डाईन्यूक्लियोटाइड कोशिकाओँ में दो संबंधित प्रकारों में पाया जाता है, एनएडीएच और एनएडीपीएच. NADP+/NADPH प्रकार अपचयी प्रतिक्रियाओं के लिये अधिक आवश्यक होता है, जबकि NAD+/NADH का प्रयोग उपचयी प्रतिक्रियाओं के लिये किया जाता है।

खनिज और सहकारक[संपादित करें]
अकार्बनिक तत्व चयापचय में महत्वपूर्ण भूमिका निभाते हैं। इनमें से कुछ (उदा.सोडियम और पोटैशियम) तो बहुतायत में पाए जाते हैं, जबकि अन्य महीन मात्राओं में काम करते हैं। स्तनपायियों के पिंड का करीब 99% भाग कार्बन, नाइट्रोजन, कैल्शियम, सोडियम, क्लोरीन, पोटैशियम, हाइड्रोजन, फास्फोरस, आक्सीजन और सल्फर तत्वों से बना होता है।[18] कार्बनिक योगिकों (प्रोटीन, वसा और कार्बोहाइड्रेट) में अधिकांशतः कार्बन और नाइट्रोजन होता है और अधिकांश आक्सीजन व हाइड्रोजन पानी में मौजूद रहते हैं।[18]
बहुतायत में मौजूद अकार्बनिक तत्व आयनीकृत इलेक्ट्रोलाइयों के रूप में काम करते हैं। सबसे महत्वपूर्ण आयन हैं, सोडियम, पोटैशियम, कैल्शियम, मैग्नीशियम, क्लोराइड, फास्फेट और कार्बनिक आयन, बाईकार्बोनेट. कोशिकाओं की झिल्लियों के पार ग्रेडियेंटों के बने रहने पर आसरण दबाव और pH बना रहता है।[19] आयन नाड़ियों और मांसपेशियों के लिये भी महत्वपूर्ण होते हैं, क्यौंकि इन ऊतकों में एक्शन पोटेंशियलें बहिर्कोशिका द्रव और कोशिका द्रव के बीच इलेक्ट्रोलाइयों के विनिमय द्वारा उत्पन्न होती हैं।[20] इलेक्ट्रोलाइट कोशिका झिल्ली के आयन चैनल नामक प्रोटीनों के जरिये कोशिकाओं के भीतर घुसते और बाहर निकलते हैं। उदा.मांस पेशी का संकुचन कोशिका झिल्ली के चैनलों और टी-नलिकाओं के जरिये कैल्शियम, सोडियम और पोटैशियम के आवागमन पर निर्भर होता है।[21]
संक्रमण धातुएं जीवों में साधारणतः ट्रेस तत्वों के रूप में मौजूद रहती हैं, जिनमें जस्ता और लोहा सबसे प्रचुर मात्रा में होते हैं।[22][23] इन धातुओं का प्रयोग कुछ प्रोटीनों में सहकारकों की तरह होता है और ये कैटालेज जैसे एंजाइमों और हीमोग्लोबिन जैसे आक्सीजन-वाहकप्रोटीनों की गतिविधि के लिये आवश्यक होते हैं।[24] ये सहकारक किसी विशिष्ट प्रोटीन से मजबूती से बंधे रहते हैं। हालांकि उत्प्रेरण के समय एंजाइम सहकारक संशोधित हो सकते हैं, उत्प्रेरण के बाद वे अपनी मूल स्थिति में लौट जाते हैं।[25][26]
अपचय[संपादित करें]
अपचय बड़े अणुओं का विघटन करने वाली चयापचयी प्रक्रियाओं का एक समूह है। इनमें भोजन कणों का विघटन और आक्सीकरण शामिल है। अपचयी प्रतिक्रियाओँ का उद्देश्य उपचयी प्रतिक्रियाओं के लिये आवश्यक ऊर्जा और पदार्थ उपलब्ध करना है। इन अपचयी प्रतिक्रियाओं की सही प्रकृति हर जीव में भिन्न होती है और जीवों को उनके ऊर्जा व कार्बन (उनके मुख्य पोषण समूह) के स्रोतों के आधार पर, नीचे दी गई सारणी के अनुसार, वर्गीकृत किया जा सकता है। कार्बनिक अणु आर्गनोट्राफों में ऊर्जा के स्रोत के रूप में प्रयोग में लाए जाते हैं, जबकि लिथोट्राफ अकार्बनिक पदार्थों का और फोटोट्राफ सूर्यप्रकाश को रसायनिक ऊर्जा के रूप में प्रयोग में लाते हैं। लेकिन, चयापचय के ये सभी प्रकार रिडाक्स प्रतिक्रियाओं पर निर्भर होते हैं, जिनमें अपघटित दानी अणुओं जैसे कार्बनिक अणुओं, पानी, अमोनिया, हाइड्रोजन सल्फाइड या फेरस आयनों से इलेक्ट्रानों का अंतरण ग्राहक अणुओं जैसे आक्सीजन, नाइट्रेट या सल्फेट में होता है।[27] पशुओं में इन प्रतिक्रियाओं में जटिल कार्बनिक अणु विघटित होकर सरलतर अणुओं जैसे कार्बन डाई आक्साइड और पानी का उत्पादन करते हैं। प्रकाश-संश्लेषक जीवों, जैसे पौधों और सायनोबैक्टीरिया में, ये इलेक्ट्रान-अंतरण प्रतिक्रियाएं ऊर्जा मुक्त नहीं करती हैं, लेकिन हमेशा सूर्यप्रकाश से अवशोषित ऊर्जा के संचयन के काम में प्रयोग की जाती हैं।[7]
-
-
- जीवों का वर्गीकरण उनके चयापचय के आधार पर
-
| ऊर्जा स्रोत | सूर्य का प्रकाश | फोटो- | -ट्रोफ | ||
| पूर्व निर्मित अणु | केमो- | ||||
| इलेक्ट्रॉन दाता | कार्बनिक यौगिक | ओर्गानो- | |||
| अकार्बनिक यौगिक | लिथो- | ||||
| कार्बन स्रोत | कार्बनिक यौगिक | हेटेरो- | |||
| अकार्बनिक यौगिक | ऑटो- | ||||
पशुओं में होने वाली सबसे आम अपचय प्रतिक्रियाएं तीन मुख्य पड़ावों में बांटी जा सकती हैं। पहले पड़ाव में, बड़े कार्बनिक अणु जैसे, प्रोटीन, पॉलिसैक्राइड या वसा पदार्थ पाचन द्वारा कोशिकाओं के बाहर उनके छोटे अंशों में बदल दिये जाते हैं। फिर, ये छोटे अणु कोशिकाओं में अवशोषित होकर और छोटे अणुओं, सामान्यतः एसिटाइल सहएंजाइम-ए (एसिटाइल-कोए) में परिणित होते हैं, जो थोड़ी ऊर्जा मुक्त करता है। अंततः, कोए का एसिटाइल समूह सिट्रिक एसिड चक्र और इलेक्ट्रान परिवहन श्रंखला में आक्सीकृत होकर पानी और कार्बन डाई आक्साइड उत्पन्न करता है, जिससे ऊर्जा मुक्त होती है, जिसे सहएंजाइम निकोटिनामाइड एडीनाइन डाईन्यूक्लियोटाइड (NAD+) के अपघटन द्वारा एनएडीएच में संचित किया जाता है।
पाचन[संपादित करें]
महाअणु जैसे स्टार्च, सेलूलोज या प्रोटीन कोशिकाओं द्वारा तेजी से अवशोषित नहीं किये जा सकते हैं और कोशिका चयापचय में उनका प्रयोग करने के पहले उन्हें छोटी इकाइयों में विघटित होना पड़ता है। कई प्रकार के एंजाइम इन पॉलिमरों को पचाते हैं। इन पाचक एंजाइमों में प्रोटीनों को अमीनो एसिडों में पचाने वाले प्रोटियेज़, पॉलिसैक्राइडों को मोनोसैक्राइडों में पचाने वाले ग्लाइकोसाइड हाइड्रोलेज़ शामिल हैं।
जीवाणु केवल अपने आस-पास पाचक एंजाइमों का स्राव करते हैं,[28][29] जबकि पशु इन एंजाइमों का सिर्फ विशेष कोशिकाओं द्वारा अपनी आंतों में स्राव करते हैं।[30] इन पराकोशिकीय एंजाइमों द्वारा मुक्त किये गए अमीनो एसिड या शर्कराएं फिर विशिष्ट सक्रिय परिवहन प्रोटीनों द्वारा कोशिकाओं में पहुंचा दी जाती हैं।[31][32]

कार्बनिक यौगिकों से ऊर्जा[संपादित करें]
कार्बोहाइड्रेट अपचय में कार्बोहाइड्रेटों को छोटी इकाइयों में विघटित किया जाता है। कार्बोहाइड्रेट मोनोसैक्राइडों में पाचन के बाद सामान्यतः कोशिकाओं में अवशोषित हो जाते हैं।[33] एक बार भीतर पहुंचने के बाद विघटन का मुख्य मार्ग ग्लाइकोलाइसिस है, जिसमें ग्लुकोज और फ्रक्टोज जैसी शर्कराएं पायरूवेट में परिणित की जाती हैं और कुछ एटीपी मुक्त होते हैं।[34] पायरूवेट कई चयापचयी मार्गों में मध्यस्थ होता है, लेकिन अधिकांश एसिटाइल-कोए में परिवर्तित हो जाता है और सिट्रिक एसिड चक्र में प्रविष्ट कर दिया जाता है। हालांकि सिट्रिक एसिड चक्र में कुछ और एटीपी उत्पन्न होता है, उसका सबसे महत्वपूर्ण उत्पादन एनएडीएच होता है, जो एसिटाइल-कोए के आक्सीकृत होने पर NAD+ से बनता है। इस आक्सीकरण से व्यर्थ उत्पाद के रूप में कार्बन डाई आक्साइड मुक्त होती है। एनएरोबिक दशाओं में, ग्लाइकालिसिस से लैक्टेट डीहाइड्रोजनेज द्वारा ग्लाइकालिसिस में पुनः प्रयोग के लिये एनएडीएच के पुनः एनएडी+ में आक्सीकरण से लैक्टेट की उत्पत्ति होती है। ग्लुकोज के विघचन का एक वैकल्पिक मार्ग पेंटोज़ फास्फेट मार्ग है, जिसमें कोएंजाइम एनएडीपीएच का अपघटन होता है और नाभिकीय अम्लों के शुगर भाग, राइबोज़ जैसी पेंटोज़ शर्कराओं का उत्पादन होता है।
वसा पदार्थ जलविच्छेदन द्वारा मुक्त वसा अम्लों और ग्लिसरॉल में अपचित होते हैं। ग्लिसरॉल ग्लाइकालिसिस में प्रवेश करता है और वसा अम्ल बीटा आक्सीकरण द्वारा विघटित होकर एसिटाइल-कोए को मुक्त करते हैं, जो सिट्रिक एसिड चक्र में काम आता है। वसा अम्ल आक्सीकृत होने पर कार्बोहाइड्रेटों की अपेक्षा अधिक ऊर्जा देते हैं क्यौंकि कार्बोहाइड्रेटों की रचनाओं में अधिक आक्सीजन होती है।
अमीनो एसिड या तो प्रोटीनों और अन्य जैवअणुओं के संश्लेषण में प्रयुक्त होते हैं, या यूरिया और कार्बन डाई आक्साइड में ऊर्जा के एक स्रोत के रूप में आक्सीकृत हो जाते हैं।[35] आक्सीकरण मार्ग का प्रारंभ किसी ट्रांसअमाइनेज द्वारा एक अमीनो समूह को हटा देने के साथ होता है। अमीनो समूह यूरिया चक्र में चला जाता है और अपने पीछे कीटो एसिड के रूप में एक विअमिनिकृत कार्बन पंजर छोड़ देता है। इस तरह के कई कीटो एसिड सिट्रिक एसिड चक्र में मध्यस्थ होते हैं, उदा. ग्लुटामेट के विअमिनीकरण से α-कीटोग्लुटारेट बनता है।[36] ग्लुकोजेनिक अमीनो एसिड भी ग्लुकोनियोजेनेसिस द्वारा ग्लुकोज में बदले जा सकते हैं। (नीचे चर्चित).[37]
ऊर्जा परिवर्तन[संपादित करें]
आक्सीकरित फास्फारिलीकरण[संपादित करें]
आक्सीकारक फास्फारिलीकरण में सिट्रिक एसिड चक्र जैसे पथों में भोजन अणुओं से निकाले गए इलेक्ट्रान आक्सीजन को अंतरित कर दिये जाते हैं और मुक्त हुई ऊर्जा का प्रयोग एटीपी बनाने के लिये किया जाता है। यह काम यूकैर्योसाइटों में इलेक्ट्रान परिवहन श्रंखला नामक प्रोटीनों द्वारा माइटोकांड्रिया की झिल्लियों में किया जाता है। प्रोकैर्योसाइटों में ये प्रोटीन कोशिका की भीतरी झिल्ली में पाए जाते हैं।[38] ये प्रोटीन अपघटित अणुओं जैसे एनएडीएच (NADH) से प्राप्त इलेक्ट्रानों को आक्सीजन पर प्रवाहित करने से उत्पन्न ऊर्जा का प्रयोग झिल्ली के पार प्रोटानों को पहुंचाने के लिये करते हैं।[39]
माइटोकांड्रिया से प्रोटानों को बाहर भेजने पर झिल्ली के पार के प्रोटान मात्रा में भिन्नता उत्पन्न हो जाती है और एक विद्युत-रसायनिक ग्रेडियेंट उत्पन्न हो जाता है।[40] यह बल प्रोटानों को वापस माइटोकांड्रिया में एटीपी (ATP) सिंथेज़ नामक एंजाइम के आधार के जरिये धकेल देता है। प्रोटानों का प्रवाह उपइकाई को घुमा देता है, जिससे सिंथेज का सक्रिय भाग अपना आकार बदल लेता है और एडीनोसीन डाईफास्फेट का फास्फारिलीकरण करके उसे एटीपी में बदल देता है।[15]
अकार्बनिक यौगिकों से ऊर्जा[संपादित करें]
कीमोलिथोट्रिप्सी प्रोकैर्योसाइटों में पाया जाने वाला एक प्रकार का चयापचय है, जिसमें अकार्बनिक यौगिकों के आक्सीकरण से ऊर्जा प्राप्त की जाती है। ये जीव हाइड्रोजन,[41] अपघटित सल्फर य़ौगिकों (जैसे सल्फाइड, हाइड्रजन सल्फाइड और थायोसल्फेट)[1], फैरस लोहे (फेल)[42] या अमोनिया[43] को अपघटन शक्ति के रूप में प्रयोग में ला सकते हैं और इन यौगिकों के आक्सीजन या नाइट्राइट जैसे इलेक्ट्रान ग्राहकों द्वारा आक्सीकरण से ऊर्जा प्राप्त करते हैं।[44] ये जीवाणु प्रक्रियाएं सर्वव्यापी जैवभूरसायनिक चक्रों जैसे एसिटोजेनेसिस, नाइट्रीकरण और विनाइट्रीकरण में महत्व रखती हैं और मिट्टी के उपजाऊपन के लिये आवश्यक होती हैं।[45][46]
प्रकाश से ऊर्जा[संपादित करें]
सूर्य के प्रकाश की ऊर्जा पौधों, सायनोबैक्टीरिया, बैंगनी बैक्टीरिया, हरे गंधक बैक्टीरिया और कुछ प्रोटिस्टों द्वारा ग्रहण की जाती है। यह प्रक्रिया, जैसा कि नीचे कहा गया है, अकसर प्रकाश-संश्लेषण के एक भाग के रूप में कार्बन डाई आक्साइड के कार्बनिक यौगिकों में परिवर्तित होने के साथ घटती है। ऊर्जा के ग्रहण करने और कार्बन का स्थिरीकरण प्रोकैर्योटों में अलग रूप से भी हो सकता है, क्यौंकि बैंगनी बैक्टीरिया और हरे गंधक बैक्टीरिया, कार्बन के स्थिरीकरण और कार्बनिक यौगिकों के किण्वन को बारी-बारी से करके सूर्य-प्रकाश को ऊर्जा के स्रोत के रूप में उपयोग में ला सकते हैं।[47][48]
कई जीवों में सूर्य की ऊर्जा को ग्रहण करने की क्रिया सैद्धांतिक रूप से आक्सीकारक फास्फारिलीकरण के समान होती है, क्यौंकि इसमें ऊर्जा प्रोटान सांद्रता ग्रेडिएंट में संचित होती है और यह प्रोटान एटीपी संश्लेषण को प्रोत्साहित करता है।[15] इस इलेक्ट्रान परिवहन श्रंखला को आगे बढ़ाने के लिये इलेक्ट्रान प्रकाश-संश्लेषण प्रतिक्रिया केंद्रों या रोडाप्सिन नामक प्रकाश-संचयी प्रोटीनों से आते हैं। प्रतिक्रिया केंद्रों को प्रकाश-संश्लेषक रंजकों के प्रकार के अनुसार दो प्रकारों में वर्गीकृत किया गया है। कई प्रकाश-संश्लेषक बैक्टीरिया में केवल एक ही प्रकार होता है, जबकि पौधों और सयानोबैक्टीरिया में दो प्रकार होते हैं।[49]
पौधों, शैवाल और सयानोबैक्टीरिया में प्रकाशतंत्र II प्रकाश ऊर्जा का प्रयोग पानी से इलेक्ट्रानों को अलग करने के लिये करता है, जिससे आक्सीजन एक व्यर्थ उत्पाद के रूप में मुक्त होती है। इसके बाद इलेक्ट्रान साइटोक्रोम b6f काम्प्लेक्स की ओर बहते हैं, जो उनकी ऊर्जा का प्रयोग क्लोरोप्लास्ट की थायलकायड झिल्ली के पार प्रोटानों को पम्प करने के लिये करते हैं।[7] ये प्रोटान पहले की तरह, एटीपी सिंथेज़ को चलाते हुए झिल्ली से वापस बाहर निकल जाते हैं। ये इलेक्ट्रान फिर प्रकाशतंत्र I मे से प्रवाहित होते हैं और कैल्विन चक्र में उपयोग के लिये सहएंजाइम एनएडीपी + के अपघटन के लिये या और एटीपी उत्पादन के लिये फिर से काम में लिये जाते हैं।[50]
उपचय[संपादित करें]
उपचय रचनात्मक चयापचयी प्रतिक्रियाओं के उस समूह को कहते हैं, जिसमें अपचय से उत्पन्न ऊर्जा को जटिल अणुओं के संश्लेषण के लिये प्रयोग में लाया जाता है। मोटे तौर पर, कोशिकीय रचना को बनाने वाले जटिल अणुओं का निर्माण छोटे और सादे अणुओं से विधिवत किया जाता है। उपचय की तीन मुख्य अवस्थाएं होती है। पहली, अमीनो एसिड, मोनोसैक्राइड, आइसोप्रेनायड और न्यूक्लियोटाइडों जैसे प्राथमिक अणुओं का उत्पादन, दूसरी, एटीपी से उर्जा का प्रयोग करके उन्हें प्रतिक्रियात्मक रूप में सक्रिय करना और तीसरी, इन प्राथमिक अणुओं को जोड़ कर जटिल अणु जैसे, प्रोटीन, पॉलिसैक्राइड, वसा पदार्थ और नाभिकीय अम्ल बनाना.
जीवों में इस बात में भिन्नता होती है, कि उनकी कोशिकाओं के कितने अणुओं का निर्माण वे स्वयं कर सकते हैं। आटोट्राफ जैसे पौधे कोशिकाओं में सरल अणुओं जौसे कार्बन डाई आक्साइड और पानी से जटिल अणुओं जैसे पॉलिसैक्राइडों और प्रोटीनों का निर्माण कर सकते हैं। दूसरी ओर, हेटेरोट्राफों को इन जटिल अणुओं के उत्पादन के लिये अधिक जटिल पदार्थों जैसे, मोनोसैक्राइडों और अमीनो एसिडों की जरूरत होती है। जीवों को उनके ऊर्जा के अंतिम स्रोत के आधार पर आगे वर्गीकृत किया जा सकता है – फोटोआटोट्राफ और फोटोहेटेरोट्राफ प्रकाश से ऊर्जा प्राप्त करते हैं, जबकि कीमोआटोट्राफ और कीमोहेटेरोट्राफ अकार्बनिक आक्सीकरण प्रतिक्रियाओं से ऊर्जा प्राप्त करते हैं।
कार्बन का स्थिरीकरण[संपादित करें]
सूर्यप्रकाश और कार्बन डाईआक्साइड (CO2) से कार्बोहाइड्रेटों के संश्लेषण को प्रकाश-संश्लेषण कहते हैं। पौधों, सयानोबैक्टीरिया और शैवाल में, आक्सीजनीय प्रकाश-संश्लेषण पानी का विच्छेद करता है, जिससे आक्सीजन व्यर्थ उत्पाद के रूप में उत्पन्न होती है। इस प्रक्रिया में, उपर्लिखित विवरण के अनुसार, प्रकाश-संश्लेषक प्रतिक्रिया केंद्रों द्वारा उत्पन्न एटीपी और एनएडीपीएच का प्रयोग CO2 को ग्लिसरेट 3-फास्फेट में बदलने के लिये किया जाता है, जिसको फिर ग्लुकोज में बदला जा सकता है। यह कार्बन-स्थिरीकरण प्रतिक्रिया कैल्विन-बेन्सन चक्र के हिस्से के रूप में एंजाइम रूबिस्को द्वारा फलीभूत की जाती है।[51] पौधों में तीन प्रकार का प्रकाश-संश्लेषण हो सकता है, सी3 कार्बन स्थिरीकरण, सी4 कारब्न स्थिरीकरण और सीएऐम प्रकाश-संश्लेषण. इनमें कैल्विन चक्र तक पहुंचने के लिये CO2 द्वारा अपनाए गए मार्ग के अनुसार भिन्नता होती है, सी3 पौधे सीधे CO2 का स्थिरीकरण करते हैं, जबकि सी4 और सीएऐम प्रकाश-संश्लेषण में तीव्र सूर्यप्रकाश और शुष्क परिस्थितियों से निपटने के लिये, सीओ2 को पहले अन्य यौगिकों में समाविष्ट किया जाता है।[52]
प्रकाश-संश्लेषक प्रोकैर्योसाइटों में कार्बन स्थिरीकरण की पद्धतियों में अधिक विविधता होती है। इसमें कार्बन डाईआक्साइड का स्थिरीकरण कैल्विन-बेन्सन चक्र, उल्टे सिट्रिक एसिड चक्र,[53] या एसिटाइल-कोए के कार्बाक्सिलीकरण द्वारा किया जा सकता है।[54][55] प्रोकैर्योटिक कीमोआटोट्राफ CO2 को कैल्विन-बेन्सन चक्र द्वारा भी स्थिर कर सकते हैं, लेकिन इस प्रतिक्रिया के लिये आवश्यक ऊर्जा अकार्बनिक यौगिकों से प्राप्त होती है।[56]
कार्बोहाइड्रेट और ग्लाइकान[संपादित करें]
कार्बोहाइड्रेट उपचय में, सरल कार्बनिक अम्लों को ग्लुकोज जैसे मोनोसैक्राइडों में बदला जा सकता है और फिर स्टार्च जैसे पलिसैक्राइडों के निर्माण के लिये प्रयोग में लाया जा सकता है। पायरूवेट, लैक्टेट, ग्लिसरॉल, ग्लिसरेट 3-फास्फेट और अमीनो एसिडों जैसे यौगिकों से ग्लुकोज के उत्पादन को ग्लुकोनियोजेनेसिस कहा जाता है। ग्लुकोलियोजेनेसिस में पायरूवेट को ग्लुकोज-6-फास्फेट में मध्यस्थों की एक श्रंखला के जरिये परिवर्तित किया जाता है, जिनमें से कई ग्लायकालिसिस में भी पाए जाते हैं।[34] लेकिन यह पथ केवल उल्टी ग्लायकालिसिस नहीं है, क्यौंकि इसके अनेक चरण गैर-ग्लायकालिटिक एंजाइमों द्वारा उत्प्रेरित किये जाते हैं। ऐसा होना महत्वपूर्ण है क्यौंकि इससे ग्लुकोज के उत्पादन और विच्छेदन के पथ के नियमन में सहायता मिलती है और दोनों पथों को किसी चक्र में एक साथ घटने से रोका जा सकता है।[57][58]
हालांकि, वसा ऊर्जा के संचय का सामान्य तरीका है, पृष्ठवंशियों जैसे मानव में इन भंडारों के वसा अम्ल ग्लुकोनियोजेनेसिस द्वारा ग्लुकोज में नहीं बदले जा सकते हैं, क्यौंकि इन जीवों में एसिटाइल-कोए को पायरूवेट में बदलने की क्षमता नहीं होती.[59] इसके लिये आवश्यक एंजाइम पोधों में होते हैं पर जानवरों में नहीं होते. फलतः लंबे समय तक बिना आहार के रहने के बाद पृष्ठवंशियों को मस्तिष्क जैसे ऊतकों, जो वसा अम्लों का चयापचय नहीं कर सकते हैं, में ग्लुकोज के स्थान पर वसा अम्लों से कीटोन कायों का उत्पादन करना पड़ता है।[60] अन्य जीवों, जैसे पौधों और बैक्टीरिया में, इस चयापचयी समस्या का समाधान ग्लयाक्सिलेट चक्र का प्रयोग करके किया जाता है, जो सिट्रिक एसिड चक्र के विकार्बाक्सीलीकरण चरण को बाईपास करके एसिटाइल-कोए को आक्जेलोएसीटेट में बदलने देती है, जिसका प्रयोग ग्लुकोज के उत्पादन के लिये किया जा सकता है।[59][61]
पॉलिसैक्राइड और ग्लाइकान विकासशील पॉलिसैक्राइड पर स्थित ग्राहक हाइड्राक्सिल समूह पर यूरिडीन डाईफास्फेट जैसे प्रतिक्रियात्मक शुगर-फास्फेट दाता से ग्लायकोसिलट्रांसफरेज द्वारा मोनोसैक्राइडों के श्रंखलात्मक जोड़ से बनाए जाते हैं। चूंकि सबस्ट्रेट के छल्ले पर स्थित कोई बी हाइड्राक्सिल समूह ग्राहक हो सकते हैं, इसलिये उत्पन्न हुए पॉलिसैक्राइडो की रचना सीधी या शाखायुक्त हो सकती है।[62] उत्पन्न पॉलिसैक्राइडों के अपने रचनात्मक या चयापचयी कर्तव्य हो सकते हैं या वे आलिगोसैकरिलट्रांसफरेजों नामक एंजाइमों द्वारा वसाओ और प्रोटीनों को अंतरित किये जा सकते हैं।[63][64]
वसा अम्ल, आइसोप्रेनायड और स्टीरायड[संपादित करें]

वसा अम्ल वसा अम्ल सिंथेज़ों द्वारा बने जाते हैं, जो एसिटाइल-कोए इकाइयों को पालिमरित करके अपघटित कर देते हैं। वसा अम्लों की एसाइल श्रंखलाएं प्रतिक्रियाओं के एक चक्र द्वारा और लंबी की जाती हैं, जो एसाइल समूह जोड़ती हैं, उसे अल्कोहल में अपघटित करती हैं, निर्जलीकरण द्वारा अल्कीन समूह में परिणित करती हैं और फिर वापस अपघटित करके अल्केन समूह में बदल देती हैं। वसा अम्ल जैवसंश्लेषण के एंजाइम दो समूहों में विभाजित किये गए हैं, पशुओं और फफूंदी में ये सभी वसा अम्ल सिंथेज प्रतिक्रियाएं एक बहुकार्यशील टाइप I प्रोटीन द्वारा फलीभूत की जाती हैं,[65] जबकि वनस्पति प्लास्टिडों और बैक्टीरिया में पृथक टाइप II एंजाइम पथमार्ग में हर चरण को पूरा करते हैं।[66][67]
टर्पीन और आइसोप्रेनायड वसाओं की एक बड़ी कक्षा हैं जिनमें कैरोटीनायड शामिल हैं और वनस्पति प्राकृतिक उत्पादनों के सबसे बड़े वर्ग का निर्माण करते हैं।[68] ये यौगिक प्रतिक्रियात्मक अणुओं आइसोपेंटेनाइल पायरोफास्फेट और डाईमेथाइलएलिल पायरोफास्फेट द्वारा दी गई आइसोप्रीन इकाइयों के जमाव और संशोधन से बनाए जाते हैं।[69] इन यौगिकों को भिन्न तरीकों से बनाया जा सकता है। पशुओं और आर्केइया में, मेवालोनेट पथमार्ग एसिटाइल-कोए से इन यौगिकों का उत्पादन करता है,[70] जबकि पौधों और बैक्टीरिया में गैर-मेवालोनेट पथमार्ग पायरूवेट और ग्लिसराल्डीहाइड 3-फास्फेट का प्रयोग करते हैं।[69][71] स्टीरायड जैवसंश्लेषण इन सक्रिय आइसोप्रीन दाताओं का प्रयोग करने वाली एक महत्वपूर्ण प्रतिक्रिया है। इसमें, आइसोप्रीन इकाइयां आपस में जुड़कर स्क्वालीन बनाती हैं और फिर दोहरी होकर छल्लों का समूह बना कर लैनास्ट्राल उत्पन्न करती हैं।[72] लैनास्ट्राल को फिर कालेस्ट्राल और अर्गोस्ट्राल जैसे अन्य स्टीरायडों में परिवर्तित किया जा सकता है।[72][73]
प्रोटीन[संपादित करें]
20 सामान्य अमीनो अम्लों के संश्लेषम की क्षमता हर जीव में भिन्न होती है। अधिकांश बैक्टीरिया और पौधे सभी बीस का संश्लेषण कर सकते हैं, लेकिन स्तनपाय़ी केवल ग्यारह अनावश्यक अमीनो अम्लों का संश्लेषण कर सकते हैं।[7] इस तरह, नौ आवश्यक अमीनो अम्ल भोजन से प्राप्त करने होते हैं। सभी अमीनो अम्ल ग्लाइकालिसिस, सिट्रिक एसिड चक्र, या पेंटोज फास्फेट पथमार्ग के मध्यस्थों से संश्लेषित किये जाते हैं। नाइट्रोजन ग्लूटामेट और ग्लूटामीन द्वारा उपलब्ध की जाती है। अमीनो अम्ल संश्लेषण उचित अल्फा-कीटो अम्ल के बनने पर निर्भर होता है, जो फिर ट्रांसअमीनीकृत होकर अमीनो अम्ल का निर्माण करता है।[74]
अमीनो एसिडों को पेप्टाइड बांडों द्वारा एक जंजीर के रूप में जोड़ कर प्रोटीनों में बदला जाता है। प्रत्येक भिन्न प्रोटीन में अमीनो एसिडों की एक अनूठी श्रंखला होती है। वर्णमाला के अक्षरों को जिस तरह जोड़ कर लगभग असीमित प्रकार के शब्द बनाए जा सकते हैं, ठीक उसी तरह अमीनो एसिडों को भी भिन्न प्रकार की श्रंखलाओं में जोड़ कर बहुत बड़ी विविधता वाले प्रोटीन बनाए जा सकते हैं। प्रोटीन उन अमीनो एसिडों से बनाए जाते हैं, जो ट्रांसफर आरएनए अणु से एक एस्टर बांड के जरिये जुड़कर सक्रिय किये गए हों. यह अमीनोएसिल-टीआरएनए प्रीकर्सर एक अमीनोएसिल टीआरएनए सिंथटेज द्वारा की गई एक एटीपी पर निर्भर प्रतिक्रिया में उत्पन्न होता है।[75] यह अमीनोएसिल-टीआरएनए तब रिबोसोम के लिये सबस्ट्रेट होता है, जो, मेसेंजर आरएनए में मौजूद श्रंखला जानकारी का प्रयोग करके लंबी होती प्रोटीन जंजीर पर अमीनो एसिड से संलग्न हो जाता है।[76]
न्यूक्लियोटाइड संश्लेषण और संग्रह[संपादित करें]
न्यूक्लियोटाइड उन पथमार्गों में अमीनो एसिडों, कार्बन डाईआक्साइड और फार्मिक एसिड से बनाए जाते हैं जिन्हें चयापचय ऊर्जा की बड़ी मात्रा में जरूरत पड़ती है।[77] फलस्वरूप, अधिकांश जीवों में पूर्वनिर्मित न्यूक्लियोटाइडों को संचित करने के लिये यथोचित व्यवस्था होती है।[77][78] प्यूरीनों का न्यूक्लियोसाइडों (रिबोसोमों से संलग्न क्षार) के रूप में संश्लेषण किया जाता है। एडीनाइन और गुआनाइन दोनों अग्रगामी न्यूक्लियोसाइड आइनोसीन मोनोफास्फेट से बनते हैं, जो अमीनो एसिडों, ग्लाइसीन, ग्लुटामीन और एस्पार्टिक एसिड से प्राप्त परमाणुओं और सहएंजाइम टेट्राहाइड्रोफोलेट से अंतरित फार्मेट का प्रयोग करके संश्लेषित किया जाता है। दूसरी ओर पायरीमिडीन, ग्लुटामीन और एस्पार्टेट से बने क्षार ओरोटेट से संश्लेषित होता है।[79]
जीनोबायोटिक और रिडाक्स चयापचय[संपादित करें]
सभी जीवों का सामना ऐसे यौगिकों से होता है, जिन्हें भोजन के रूप में प्रयोग में नहीं लाया जा सकता है और जो यदि कोशिकाओं में जमा हो जाएं तो हानिकारक हो सकते हैं क्यौंकि उनकी कोई चयापचयी भूमिका नहीं होती. ऐसे हानिकारक यौगिकों को यीनोबायोटिक कहा जाता है।[80] संश्लेषित औषधियों, प्राकृतिक विषों और एंटीबायोटिकों जैसे जीनोबयोटिकों को जीनोबायोटिक-चयापचयी एंजाइमों के एक समूह द्वारा निष्क्रिय किया जाता है। मनुष्यों में, इनमें साइटोक्रोम पी450 आक्सिडेज,[81] यूडीपी-ग्लुकुरुनोसिलट्रांसफरेज,[82] और ग्लुटाथयोन S -ट्रांसफरेज शामिल हैं।[83] एंजाइमों का यह तंत्र तीन अवस्थाओं में कार्य करता है, पहले जीनोबायोटिक को आक्सीकृत करना (पहली अवस्था) और फिर जल-घुलनशील समूहों को अणु पर कान्जुगेट (दूसरी अवस्था) करना. संशोधित जल-घुलनशील जीनोबायोटिक को फिर कोशिका के बाहर पम्प कर दिया जाता है और बहुकोशिकीय जीवों में बाहर निकालने के पहले और चयपचयित किया जाता है। इकालाजी में ये प्रतिक्रियाएं दूषक तत्वों के जीवाणुओं द्वारा जैवअपघटन और दूषित जमीन व तेल के रिस जाने पर जैवउपचार के लिये विशेषकर महत्वपूर्ण हैं।[84] इनमें से कई जीवाणु प्रतिक्रियाएं बहुकोशिकीय जीवों में भी होती हैं, लेकिन जीवाणुओं के अविश्वसनीय विविध प्रकारों के कारण ये जीव बहुकोशिकीय जीवों की अपेक्षा कहीं अधिक प्रकार के जीनोबायोटिकों का सामना कर सकते हैं और आर्गैनोक्लोराइड यौगिकों जैसे हठी कार्बनिक दूषकों से भी निपट सकते हैं।[85]
एयरोबिक जीवों से संबंधित एक समस्या है, आक्सीकरण दबाव.[86] इसमें, आक्सीकरणीय फास्फारिलीकरण और प्रोटीनों के दोहरेपन के समय डाईसल्फाइड बांडों के निर्माण सहित प्रक्रियाएं हाइड्रोजन पराक्साइड जैसी प्रतिक्रियात्मक जातियों का उत्पादन करती हैं।[87] ये हानिकारक आक्सीडैंट आक्सीकरणविरोधी चयापचयकों जैसे ग्लूटाथयोन और एंजाइमों जैसे कैटालेजों और पराक्सिडेजों द्वारा निष्कासित किये जाते हैं।[88][89]
जीवित जन्तुओं की ऊष्मप्रगैतिकी[संपादित करें]
जीवित जन्तुओं को ऊष्मप्रगैतिकी के नियमों का पालन करना आवश्यक होता है, जो ऊष्मा के अंतरण और कार्य के बारे में बतलाते हैं। ऊष्मप्रगैतिकी के दूसरे नियम के अनुसार, किसी भी बंद तंत्र में एंट्रापी (विकार) में वृद्धि होती है। हालांकि जीवित जंतुओं की आश्चर्य़पूर्ण जटिलता इस नियम के विरूद्ध जाती है, जीवन संभव है क्यौंकि सभी जीव खुले तंत्र हैं जो अपने आस-पास के वातावरण से पदार्थ और ऊर्जा का विनिमय करते हैं। इस तरह जीवित तंत्र संतुलन में नहीं होते, बल्कि नष्ट होने वाले तंत्र हैं जो अपने पर्यावरणों में एंट्रापी में अधिक वृद्धि करके अपनी उच्च जटिलता की स्थिति बने रखते हैं।[90] कोशिका का चयापचय इसे अपचय की स्वाभाविक प्रक्रियाओं को उपचय की अस्वाभाविक प्रक्रियाओं से युग्मित करके संभव करता है। ऊष्मप्रगैतिकी की भाषा में, चयापचय असंतुलन उत्पन्न करके संतुलन बनाए रखता है।[91]
नियमन और नियंत्रण[संपादित करें]
चूंकि अधिकांश जीवों के पर्यावरण लगातार बदलते रहते हैं, इसलिये चयापचयी प्रतिक्रियाओं का कोशिकाओं में एक स्थिर दशा बनाए रखने के लिये बारीकी से नियमित होना आवश्यक है, जिसे होमियोस्टैसिस कहते हैं।[92][93] चयापचयी नियमन जीवों को संकेतों के प्रति जवाब देने और अपने पर्यावरणों से सक्रिय रूप से अंतर्क्रिया करने में सहायक होते हैं।[94] चयापचयी पथमार्गों के नियंत्रण की क्रिया को समझने के लिये दो आपस में मजबूती से जुड़े सिद्धांत महत्वपूर्ण हैं। एक, किसी पथमार्ग में एंजाइम के नियमन के अनुसार संकेत के प्रति उसकी गतिविधि बढ़ती या घटती है। दूसरे, इस एंजाइम द्वारा किया गया नियंत्रण ही पथमार्ग की कुल दर पर गतिविधि में हुए परिवर्तनों का प्रभाव है। (पथमार्ग द्वारा बहाव)[95] उदा.एंजाइम अपनी गतिविधि में बड़े परिवर्तन दिखाता है (अर्थात् बड़े तौर पर नियमित होता है), लेकिन यदि इन परिवर्तनों का चयापचयी पथमार्ग के बहाव पर थोड़ा सा प्रभाव हो, तो यह एंजाइम पथमार्ग के नियंत्रण में शामिल नहीं है।[96]

चयापचय नियमन के कई स्तर होते हैं। आंतरिक नियमन में चयापचयी पथमार्ग स्वतःनियमन करके सबस्ट्रेटों या उत्पादनों के स्तरों में परिवर्तनों के प्रति प्रतिक्रिया करता है। उदा.उत्पादन की मात्रा में कमी होने पर पथमार्ग से बहाव में वृद्धि हो जाती है।[95] इस तरह के नियमन में अकसर पथमार्ग के अनेक एंजाइमों की गतिविधियों का एलोस्टेरिक नियमन होता है।[97] बाह्य नियंत्रण में बहुकोशिकीय जीव की एक कोशिका अन्य कोशिकाओं के संकेतों के अनुसार अपने चयापचय में परिवर्तन लाती हैं। ये संकेत सामान्यतः हारमोनों और विकास कारकों जैसे घुलनशील संदेशवाहकों के रूप में होते हैं और कोशिका-सतह पर विशिष्ट ग्राहकों द्वारा पहचाने जाते हैं।[98] फिर ये संकेत कोशिका के भीतर द्वितीय संदेशवाहक तंत्रों द्वारा संचरित किये जाते हैं, जो अकसर प्रोटीनों के फास्फारिलीकरण में लगे होते हैं।[99]
बाह्य नियंत्रण का एक बहुत अच्छी तरह से समझा गया उदाहरण है, इन्सुलिन हारमोन द्वारा ग्लुकोज चयापचय का नियमन.[100] इन्सुलिन का उत्पादन रक्त ग्लुकोज स्तरों के बढ़ने पर होता है। कोशिकाओं पर स्थित इन्सुलिन ग्राहकों से हारमोन के जुड़ने पर प्रोटीन काइनेजों का प्रपात सक्रिय हो जाता है, जो कोशिकाओं द्वारा ग्लुकोज लेकर उसे वसा अम्लों और ग्लायकोजन जैसे संचय अणुओं में परिवर्तित करवाता है।[101] ग्लायकोजन का चयापचय एंजाइम फास्फारिलेज, जो ग्लायकोजन का विघटन करता है और ग्लायकोजन सिंथेज, जो उसे बनाता है, द्वारा नियंत्रित होता है। फास्फारिलीकरण ग्लायकोजन सिंथेज का अवरोध करता है, लेकिन फास्फारिलेज को सक्रिय करता है। इन्सुलिन प्रोटीन फास्फेटेजों को सक्रिय करके और इन एंजाइमों के फास्फारिलीकरण में कमी लाकर ग्लायकोजन का संश्लेषण करवाता है।[102]
विकास[संपादित करें]

उपर्लिखित चयापचय के केंद्रीय पथमार्ग, जैसे ग्लायकालिसिस औऱ सिट्रिक एसिड चक्र, जीवित वस्तुओं के तीनों वर्गों में होते हैं और पिछले विश्व पूर्वज में मौजूद थे।[3][103] यह सार्वभौमिक पूर्वज कोशिका प्रोकार्योटिक और शायद मेथेनोजन थी जिसमें व्यापक अमीनो एसिड, न्यूक्लियोटाइड, कार्बोहाइड्रेट और वसा चयापचय होता था।[104][105] इन प्राचीन पथमार्गों का आगे के विकास में रखा जाना उनकी विशिष्ट चयापचयी समस्याओं के लिये इन प्रतिक्रियाओं का उचित समाधान होना संभव है, क्यौंकि ग्लायकालिसिस और सिट्रिक एसिड चक्र जैसे पथमार्ग बड़े यथोचित रूप से और कम से कम चरणों में उनके अंत-उत्पादों का उत्पादन करते हैं।[4][5] एंजाइम पर आधारित चयापचय के पहले पथमार्ग प्यूरीन न्यूक्लियोटाइड चयापचय के हिस्से हो सकते हैं, जिसमें पहले के चयापचयी पथमार्ग प्राचीन आरएनए दुनिया के भाग थे।[106]
नए चयापचयी पथमार्गों के उत्पन्न होने के तरीकों को समझाने के लिये कई माडल प्रस्तुत किये गए हैं। इनमें नए एंजाइमों का किसी छोटे पूर्वज पथमार्ग से श्रंखला में जुड़ना, सारे पथमार्गों के प्रतिरूप बनाकर फिर उनका हट जाना, पहले से मौजूद एजाइमों का चयन और नवीन प्रतिक्रिया पथमार्ग में उनका जमाव शामिल है।[107] इन प्रक्रियाओं का अपेक्षात्मक महत्व स्पष्ट नहीं है, लेकिन जीनोमिक अध्ययनों के अनुसार पथमार्ग के एंजाइमों के साझा पूर्वज होते हैं, जिससे ऐसा लगता है कि कई पथमार्ग बारी-बारी से उत्पन्न हुए हैं, जिनमें पथमार्ग में पहले से मौजूद चरणों में नए कार्य-कलाप बनते हैं।[108] चयापचयी नेटवर्क में प्रोटीनों की रचनाओं के विकास के लिये किये गए अध्ययनों से प्राप्त एक वैकल्पिक माडल के अनुसार एंजाइमों का चयन व्यापक रूप से होता है (मैनेट डेटाबेस में स्पष्ट है),[109] जिसमें भिन्न चयापचयी पथमार्गों में समान प्रकार के कार्य करने के लिये एंजाइम उधार लिये जाते हैं।[110] इन चयन प्रक्रियाओं के कारण एक विकासीय एंजाइमेटिक मोजैक बनता है। एक तीसरी संभावना है, चयापचय के कुछ भाग माड्यूलों की तरह रह सकते हैं, जिन्हें भिन्न पथमार्गों में पुनः काम में लिया जा सकता है और जो भिन्न अणुओं में समान तरह के कार्य करते हैं।[111]
नए चयापचयी पथमार्गों के विकास की तरह, विकास के कारण चयापचयी कार्यशीलता में कमी आ सकती है। उदा. कुछ परजीवियों में जीवन के लिये अनावश्यक चयापचयी प्रक्रियाएं नहीं होती हैं और पहले से बने हुए अमीनो एसिड, न्यूक्लियोटाइड और कार्बोहाइड्रेट मेजबान द्वारा खा लिये जाते हैं।[112] ऐसी ही चयापचयी क्षमताओं में कमी एंडोसिम्बयाटिक जीवों में देखी जाती है।[113]
जांच और परिवर्तन[संपादित करें]

चयापचय का अध्ययन मान्य रूप से अपघटीय तरीके से किया जाता है, जो एक चयापचय पथमार्ग पर केंद्रित होता है। इसमें सबसे महत्वपूर्ण है, सम्पूर्ण जीव, ऊतक और कोशिकीय स्तर पर रेडियोसक्रिय लेसरों का प्रयोग, जो रेडियोसक्रिय रूप से लेबल किये गए मध्यस्थों और उत्पादनों को पहचान कर पूर्वजों से लेकर अंतिम उत्पादन तक के पथमार्गों को परिभाषित करते हैं।[114] इन रसायनिक प्रतिक्रियाओं को उत्प्रेरित करने वाले एंजाइमों का तब शुद्धीकरण किया जा सकता है और उनकी गतिकी व अवरोधकों के प्रति उनकी प्रतिक्रियाओं की जांच की जा सकती है। एक समानांतर तरीका है, कोशिका या ऊतक में छोटे अणुओं को पहचानना. इन अणुओं के एक पूर्ण समूह को मेटाबोलोम कहा जाता है। कुल मिला कर इन अध्ययनों से सरल चयापचयी पथमार्गों की रचना और कार्य के बारे में अच्छी जानकारी मिलती है, लेकिन अधिक जटिल तंत्रों जैसे संपूर्ण कोशिका के चयापचय पर उन्हें लागू करने पर अपर्याप्त लगते हैं।[115]
विभिन्न प्रकार के हजारों एंजाइमों से युक्त कोशिकाओं के चयापचयी जाल की जटिलता का अंदाजा दांयी ओर दिये गए चित्र से लगाया जा सकता है, जिसमें सिर्फ 43 प्रोटीनों और 40 चयापचकों के बीच अंतर्क्रुया को दर्शाया गया है – जीनोमों की श्रंखलाएं 45000 जीनों तक की फेहरिस्त उपलब्ध करती है।[116] लेकिन अब इस जीनोमिक जानकारी का प्रयोग करके रसायनिक प्रतिक्रियाओं के संपूर्ण जालों का पुनर्निर्माण और उनके बर्ताव को समझने के लिये अधिक पूर्ण गणितीय माडल बनाना संभव है।[117] ये माडल विशेष रूप से शक्तिशाली तब होते हैं जब उनका प्रयोग प्रोटीयोमिक और डीएनए माइक्रोऐरे अध्ययनों से प्राप्त जीन एक्सप्रेशन विषयक जानकारी को मान्य तरीकों से प्राप्त पथमार्ग और चयापचयी जानकारी से एकीकृत करने के लिये किया जाता है।[118] इन तकनीकों का प्रयोग करके, मानव चयापचय का एक माडल बनाया गया है, जो भविष्य में औषधि की खोज और जैवरसायनिक शोध का मार्गदर्शन करेगा.[119] ये माडल अभी नेटवर्क विश्लेषण में समान प्रोटीनों या चयापचयकों वाले समूहों में मानवी रोगों के वर्गीकरण के लिये प्रयोग में लाए जा रहे हैं।[120][121]
बैक्टीरिया के चयापचयी नेटवर्क बो-टाई[122][123][124] संयोजन का अच्छा उदाहरण लगते हैं, जो अपेक्षाकृत कम मध्यस्थ मुद्राओं का प्रयोग करके पोषकों की बड़ी श्रंखलाओं की सहायता से बड़ी विविधता वाले उत्पादों और जटिल महाअणुओं को उत्पन्न कर सकते हैं।
इस जानकारी का एक मुख्य तकनीकी उपयोग चयापचयी इंजीनियरिंग है। इसमें खमीर, वनस्पति या बैक्टीरिया जैसे जीव जीनों में संशोधन द्वारा उन्हें जैवतकनीकी में अधिक उपयोगी और एंटीबायोटिकों जैसी औषधियों या 1,3-प्रोपेनडयाल और शिकिमिक एसिड जैसे औद्यौगिक रसायनों के उत्पादन में मददगार बनाया जाता है।[125] इन जीनीय संशोधनों का उद्देश्य उत्पादन में लगने वाली ऊर्जा की मात्रा को कम करने और व्यर्थ पदार्थों का उत्पादन कम करने के लिये किया जाता है।[126]
इतिहास[संपादित करें]

मेटाबोलिज्म (चयापचय) शब्द की उत्पत्ति ग्रीक शब्द, मेटाबोलिस्मॉस – परिवर्तन या उलट देना – से हुई है।[127] चयापचय के वैज्ञानिक अध्ययन का इतिहास कई शताब्दियों पुराना है और प्रारंभिक अध्ययनों में संपूर्ण पशुओं की परीक्षा से लेकर, आधुनिक जैवरसायनशास्त्र में व्यक्तिगत चयापचयी प्रतिक्रियाओं की जांच तक फैला है। चयापचय का सिद्धांत इब्न अल-नफीस (1213-1288) के समय से है, जिसने बताया कि, ‘शरीर और उसके भाग लगातार विघटन और पोषण की स्थिति में रहते हैं।[128] मानव के चयापचय के पहले प्रयोगों का प्रकाशन सैंटोरियो सैंटोरियो ने 1614 में उनकी पुस्तक आर्स डी स्टैटिका मेडेसिना में किया।[129] उसने बताया कि कैसे उसने अपने आपको भोजन करने, सोने, काम करने, मैथुन, उपवास, पीने और मलत्याग करने के पहले और बाद तौला. उसने पाया कि उसके द्वारा लिये गए आहार का अधिकांश भाग ‘असंवेदी स्वेदन’ के जरिये गायब हो गया।
इन प्रारंभिक अध्ययनों में, इन चयापचयी प्रक्रियाओं के तरीकों को पहचाना नहीं गया है और यह समझा जाता था कि कोई दैवी शक्ति जीवित ऊतक को नियंत्रित करती है।[130] 19वीं शताब्दी में खमीर द्वारा शक्कर के अल्कोहल में किण्वन का अध्ययन करते समय, लुई पास्चर ने देखा कि किण्वन का उत्प्रेरण खमीर कोशिकाओं में स्थित पदार्थों द्वारा किया जाता है, जिन्हें उसने ‘किण्वक’ का नाम दिया. उसने लिखा कि, ’अल्कोहली किण्वन खमीर कोशिकाओं के जीवन और संयोजन से संबंधित एक कार्य है और इसका कोशिकाओं की मृत्यु या सड़ने से कोई संबंध नहीं है’.[131] इस खोज और फ्रेड्रिच वोह्लर द्वारा 1828 में यूरिया के रसायनिक संश्लेषण के प्रकाशन से यह सिद्ध हुआ कि कोशिकाओं में पाए जाने वाले कार्बनिक यौगिकों और रसायनिक प्रतिक्रियाओं और रसायनशास्त्र के अन्य किसी भी भाग में सैद्धांतिक रूप से कोई भिन्नता नहीं है।[132]
20वीं शताब्दी के शुरू में एड्वर्ड बकनर द्वारा एंजाइमों की खोज के बाद चयापचय की रसायनिक प्रतिक्रियाओं और कोशिकाओं के जीववैज्ञानिक अध्ययन अलग से किये जाने लगे और जैवरसायनशास्त्र की शुरूआत हुई.[133] प्रारंभिक 20वीं शताब्दी में जैवरसायनिक जानकारी तेजी से बढ़ी. इन आधुनिक जैवरसायनज्ञों में सबसे सक्रिय थे हांस क्रेब्स, जिन्होंने चयापचय के अध्ययन में बड़ा योगदान किया।[134] उन्होंने यूरिया चक्र और हांस कार्नबर्ग के साथ काम करते हुए, सिट्रिक एसिड चक्र और ग्लयाक्सिलेट चक्र का आविष्कार किया।[135][61] आधुनिक जैवरसायनिक शोध को नई तकनीकों जैसे, क्रोमेटोग्राफी, एक्सरे डाइफ्रैक्शन, एनएमआर स्पेक्ट्रोस्कोपी, रेडियोआइसोटोपिक लेबलीकरण, इलेक्ट्रान माइक्रोस्कोपी और आण्विक गतिकी सिमुलेशन से बहुत सहायता मिली है। इन तकनीकों से कोशिकाओं में अनेक अणुओं और चयापचयी पथमार्गों की खोज और विस्तृत विश्लेषण संभव हुआ है।
इन्हें भी देखें[संपादित करें]
| विकिपुस्तक पर चयापचय से सम्बन्धित अधिक जानकारी है। |
| विकिविश्वविद्यालय में Topic:Biochemistry पर पाठ्य सामग्री उपलब्ध है: |
- ऐनथ्रोपोजेनिक चयापचय
- आधारिक चयापचय दर
- कैलोरीमेट्री
- चयापचय की अंतर्जात त्रुटि
- लोहे-सल्फर दुनिया सिद्धांत, "चयापचय पहले" मूल के जीवन का सिद्धांत.
- रेस्पिरोमेट्री
- भोजन की थेर्मिक प्रभाव
- पानी चयापचय
- सल्फर चयापचय
- ऐंटीमेटाबोलाईट
सन्दर्भ[संपादित करें]
- ↑ अ आ Friedrich C (1998). "Physiology and genetics of sulfur-oxidizing bacteria". Adv Microb Physiol. 39: 235–89. PMID 9328649. डीओआइ:10.1016/S0065-2911(08)60018-1.
- ↑ Pace NR (2001). "The universal nature of biochemistry". Proc. Natl. Acad. Sci. U.S.A. 98 (3): 805–8. PMID 11158550. डीओआइ:10.1073/pnas.98.3.805. पी॰एम॰सी॰ 33372. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद) - ↑ अ आ Smith E, Morowitz H (2004). "Universality in intermediary metabolism". Proc Natl Acad Sci USA. 101 (36): 13168–73. PMID 15340153. डीओआइ:10.1073/pnas.0404922101. पी॰एम॰सी॰ 516543. मूल से 2 जून 2020 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ अ आ Ebenhöh O, Heinrich R (2001). "Evolutionary optimization of metabolic pathways. Theoretical reconstruction of the stoichiometry of ATP and NADH producing systems". Bull Math Biol. 63 (1): 21–55. PMID 11146883. डीओआइ:10.1006/bulm.2000.0197.
- ↑ अ आ Meléndez-Hevia E, Waddell T, Cascante M (1996). "The puzzle of the Krebs citric acid cycle: assembling the pieces of chemically feasible reactions, and opportunism in the design of metabolic pathways during evolution". J Mol Evol. 43 (3): 293–303. PMID 8703096. डीओआइ:10.1007/BF02338838.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Michie K, Löwe J (2006). "Dynamic filaments of the bacterial cytoskeleton". Annu Rev Biochem. 75: 467–92. PMID 16756499. डीओआइ:10.1146/annurev.biochem.75.103004.142452.
- ↑ अ आ इ ई उ ऊ ए Nelson, David L.; Michael M. Cox (2005). Lehninger Principles of Biochemistry. New York: W. H. Freeman and company. पपृ॰ 841. आई॰ऍस॰बी॰ऍन॰ 0-7167-4339-6. सन्दर्भ त्रुटि:
<ref>अमान्य टैग है; "Nelson" नाम कई बार विभिन्न सामग्रियों में परिभाषित हो चुका है - ↑ Fahy E, Subramaniam S, Brown H, Glass C, Merrill A, Murphy R, Raetz C, Russell D, Seyama Y, Shaw W, Shimizu T, Spener F, van Meer G, VanNieuwenhze M, White S, Witztum J, Dennis E (2005). "A comprehensive classification system for lipids". J Lipid Res. 46 (5): 839–61. PMID 15722563. डीओआइ:10.1194/jlr.E400004-JLR200. मूल से 24 अगस्त 2010 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ "Nomenclature of Lipids". IUPAC-IUB Commission on Biochemical Nomenclature (CBN). मूल से 16 फ़रवरी 2007 को पुरालेखित. अभिगमन तिथि 2007-03-08.
- ↑ Hegardt F (1999). "Mitochondrial 3-hydroxy-3-methylglutaryl-CoA synthase: a control enzyme in ketogenesis". Biochem J. 338 (Pt 3): 569–82. PMID 10051425. डीओआइ:10.1042/0264-6021:3380569. पी॰एम॰सी॰ 1220089.
- ↑ Raman R, Raguram S, Venkataraman G, Paulson J, Sasisekharan R (2005). "Glycomics: an integrated systems approach to structure-function relationships of glycans". Nat Methods. 2 (11): 817–24. PMID 16278650. डीओआइ:10.1038/nmeth807.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Sierra S, Kupfer B, Kaiser R (2005). "Basics of the virology of HIV-1 and its replication". J Clin Virol. 34 (4): 233–44. PMID 16198625. डीओआइ:10.1016/j.jcv.2005.09.004.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ अ आ Wimmer M, Rose I (1978). "Mechanisms of enzyme-catalyzed group transfer reactions". Annu Rev Biochem. 47: 1031–78. PMID 354490. डीओआइ:10.1146/annurev.bi.47.070178.005123.
- ↑ Mitchell P (1979). "The Ninth Sir Hans Krebs Lecture. Compartmentation and communication in living systems. Ligand conduction: a general catalytic principle in chemical, osmotic and chemiosmotic reaction systems". Eur J Biochem. 95 (1): 1–20. PMID 378655. डीओआइ:10.1111/j.1432-1033.1979.tb12934.x.
- ↑ अ आ इ ई Dimroth P, von Ballmoos C, Meier T (2006). "Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series". EMBO Rep. 7 (3): 276–82. PMID 16607397. डीओआइ:10.1038/sj.embor.7400646. पी॰एम॰सी॰ 1456893. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Coulston, Ann; Kerner, John; Hattner, JoAnn; Srivastava, Ashini (2006). "Nutrition Principles and Clinical Nutrition". Stanford School of Medicine Nutrition Courses. SUMMIT.
- ↑ Pollak N, Dölle C, Ziegler M (2007). "The power to reduce: pyridine nucleotides—small molecules with a multitude of functions". Biochem J. 402 (2): 205–18. PMID 17295611. डीओआइ:10.1042/BJ20061638. पी॰एम॰सी॰ 1798440.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ अ आ Heymsfield S, Waki M, Kehayias J, Lichtman S, Dilmanian F, Kamen Y, Wang J, Pierson R (1991). "Chemical and elemental analysis of humans in vivo using improved body composition models". Am J Physiol. 261 (2 Pt 1): E190–8. PMID 1872381.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Sychrová H (2004). "Yeast as a model organism to study transport and homeostasis of alkali metal cations" (PDF). Physiol Res. 53 Suppl 1: S91–8. PMID 15119939. मूल (PDF) से 25 अक्तूबर 2011 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Levitan I (1988). "Modulation of ion channels in neurons and other cells". Annu Rev Neurosci. 11: 119–36. PMID 2452594. डीओआइ:10.1146/annurev.ne.11.030188.001003.
- ↑ Dulhunty A (2006). "Excitation-contraction coupling from the 1950s into the new millennium". Clin Exp Pharmacol Physiol. 33 (9): 763–72. PMID 16922804. डीओआइ:10.1111/j.1440-1681.2006.04441.x.
- ↑ Mahan D, Shields R (1998). "Macro- and micromineral composition of pigs from birth to 145 kilograms of body weight". J Anim Sci. 76 (2): 506–12. PMID 9498359. मूल से 30 अप्रैल 2011 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Husted S, Mikkelsen B, Jensen J, Nielsen N (2004). "Elemental fingerprint analysis of barley (Hordeum vulgare) using inductively coupled plasma mass spectrometry, isotope-ratio mass spectrometry, and multivariate statistics". Anal Bioanal Chem. 378 (1): 171–82. PMID 14551660. डीओआइ:10.1007/s00216-003-2219-0.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Finney L, O'Halloran T (2003). "Transition metal speciation in the cell: insights from the chemistry of metal ion receptors". Science. 300 (5621): 931–6. PMID 12738850. डीओआइ:10.1126/science.1085049.
- ↑ Cousins R, Liuzzi J, Lichten L (2006). "Mammalian zinc transport, trafficking, and signals". J Biol Chem. 281 (34): 24085–9. PMID 16793761. डीओआइ:10.1074/jbc.R600011200. मूल से 5 नवंबर 2008 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Dunn L, Rahmanto Y, Richardson D (2007). "Iron uptake and metabolism in the new millennium". Trends Cell Biol. 17 (2): 93–100. PMID 17194590. डीओआइ:10.1016/j.tcb.2006.12.003.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Nealson K, Conrad P (1999). "Life: past, present and future". Philos Trans R Soc Lond B Biol Sci. 354 (1392): 1923–39. PMID 10670014. डीओआइ:10.1098/rstb.1999.0532. पी॰एम॰सी॰ 1692713. मूल से 17 मार्च 2020 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Häse C, Finkelstein R (1993). "Bacterial extracellular zinc-containing metalloproteases". Microbiol Rev. 57 (4): 823–37. PMID 8302217. पी॰एम॰सी॰ 372940. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद) - ↑ Gupta R, Gupta N, Rathi P (2004). "Bacterial lipases: an overview of production, purification and biochemical properties". Appl Microbiol Biotechnol. 64 (6): 763–81. PMID 14966663. डीओआइ:10.1007/s00253-004-1568-8.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Hoyle T (1997). "The digestive system: linking theory and practice". Br J Nurs. 6 (22): 1285–91. PMID 9470654.
- ↑ Souba W, Pacitti A (1992). "How amino acids get into cells: mechanisms, models, menus, and mediators". JPEN J Parenter Enteral Nutr. 16 (6): 569–78. PMID 1494216. डीओआइ:10.1177/0148607192016006569.
- ↑ Barrett M, Walmsley A, Gould G (1999). "Structure and function of facilitative sugar transporters". Curr Opin Cell Biol. 11 (4): 496–502. PMID 10449337. डीओआइ:10.1016/S0955-0674(99)80072-6.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Bell G, Burant C, Takeda J, Gould G (1993). "Structure and function of mammalian facilitative sugar transporters". J Biol Chem. 268 (26): 19161–4. PMID 8366068.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ अ आ Bouché C, Serdy S, Kahn C, Goldfine A (2004). "The cellular fate of glucose and its relevance in type 2 diabetes". Endocr Rev. 25 (5): 807–30. PMID 15466941. डीओआइ:10.1210/er.2003-0026. मूल से 4 दिसंबर 2012 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Sakami W, Harrington H (1963). "Amino acid metabolism". Annu Rev Biochem. 32: 355–98. PMID 14144484. डीओआइ:10.1146/annurev.bi.32.070163.002035.
- ↑ Brosnan J (2000). "Glutamate, at the interface between amino acid and carbohydrate metabolism". J Nutr. 130 (4S Suppl): 988S–90S. PMID 10736367. मूल से 28 फ़रवरी 2007 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Young V, Ajami A (2001). "Glutamine: the emperor or his clothes?". J Nutr. 131 (9 Suppl): 2449S–59S, discussion 2486S–7S. PMID 11533293. मूल से 25 फ़रवरी 2007 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Hosler J, Ferguson-Miller S, Mills D (2006). "Energy transduction: proton transfer through the respiratory complexes". Annu Rev Biochem. 75: 165–87. PMID 16756489. डीओआइ:10.1146/annurev.biochem.75.062003.101730. पी॰एम॰सी॰ 2659341.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Schultz B, Chan S (2001). "Structures and proton-pumping strategies of mitochondrial respiratory enzymes". Annu Rev Biophys Biomol Struct. 30: 23–65. PMID 11340051. डीओआइ:10.1146/annurev.biophys.30.1.23.
- ↑ Capaldi R, Aggeler R (2002). "Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor". Trends Biochem Sci. 27 (3): 154–60. PMID 11893513. डीओआइ:10.1016/S0968-0004(01)02051-5.
- ↑ Friedrich B, Schwartz E (1993). "Molecular biology of hydrogen utilization in aerobic chemolithotrophs". Annu Rev Microbiol. 47: 351–83. PMID 8257102. डीओआइ:10.1146/annurev.mi.47.100193.002031.
- ↑ Weber K, Achenbach L, Coates J (2006). "Microorganisms pumping iron: anaerobic microbial iron oxidation and reduction". Nat Rev Microbiol. 4 (10): 752–64. PMID 16980937. डीओआइ:10.1038/nrmicro1490.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Jetten M, Strous M, van de Pas-Schoonen K, Schalk J, van Dongen U, van de Graaf A, Logemann S, Muyzer G, van Loosdrecht M, Kuenen J (1998). "The anaerobic oxidation of ammonium". FEMS Microbiol Rev. 22 (5): 421–37. PMID 9990725. डीओआइ:10.1111/j.1574-6976.1998.tb00379.x.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Simon J (2002). "Enzymology and bioenergetics of respiratory nitrite ammonification". FEMS Microbiol Rev. 26 (3): 285–309. PMID 12165429. डीओआइ:10.1111/j.1574-6976.2002.tb00616.x.
- ↑ Conrad R (1996). "Soil microorganisms as controllers of atmospheric trace gases (H2, CO, CH4, OCS, N2O, and NO)". Microbiol Rev. 60 (4): 609–40. PMID 8987358. पी॰एम॰सी॰ 239458.
- ↑ Barea J, Pozo M, Azcón R, Azcón-Aguilar C (2005). "Microbial co-operation in the rhizosphere". J Exp Bot. 56 (417): 1761–78. PMID 15911555. डीओआइ:10.1093/jxb/eri197. मूल से 16 फ़रवरी 2007 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ van der Meer M, Schouten S, Bateson M, Nübel U, Wieland A, Kühl M, de Leeuw J, Sinninghe Damsté J, Ward D (2005). "Diel variations in carbon metabolism by green nonsulfur-like bacteria in alkaline siliceous hot spring microbial mats from Yellowstone National Park". Appl Environ Microbiol. 71 (7): 3978–86. PMID 16000812. डीओआइ:10.1128/AEM.71.7.3978-3986.2005. पी॰एम॰सी॰ 1168979. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)[मृत कड़ियाँ] - ↑ Tichi M, Tabita F (2001). "Interactive control of Rhodobacter capsulatus redox-balancing systems during phototrophic metabolism". J Bacteriol. 183 (21): 6344–54. PMID 11591679. डीओआइ:10.1128/JB.183.21.6344-6354.2001. पी॰एम॰सी॰ 100130.
- ↑ Allen J, Williams J (1998). "Photosynthetic reaction centers". FEBS Lett. 438 (1–2): 5–9. PMID 9821949. डीओआइ:10.1016/S0014-5793(98)01245-9.
- ↑ Munekage Y, Hashimoto M, Miyake C, Tomizawa K, Endo T, Tasaka M, Shikanai T (2004). "Cyclic electron flow around photosystem I is essential for photosynthesis". Nature. 429 (6991): 579–82. PMID 15175756. डीओआइ:10.1038/nature02598.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Miziorko H, Lorimer G (1983). "Ribulose-1,5-bisphosphate carboxylase-oxygenase". Annu Rev Biochem. 52: 507–35. PMID 6351728. डीओआइ:10.1146/annurev.bi.52.070183.002451.
- ↑ Dodd A, Borland A, Haslam R, Griffiths H, Maxwell K (2002). "Crassulacean acid metabolism: plastic, fantastic". J Exp Bot. 53 (369): 569–80. PMID 11886877. डीओआइ:10.1093/jexbot/53.369.569. मूल से 25 जनवरी 2009 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Hügler M, Wirsen C, Fuchs G, Taylor C, Sievert S (2005). "Evidence for autotrophic CO2 fixation via the reductive tricarboxylic acid cycle by members of the epsilon subdivision of proteobacteria". J Bacteriol. 187 (9): 3020–7. PMID 15838028. डीओआइ:10.1128/JB.187.9.3020-3027.2005. पी॰एम॰सी॰ 1082812. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Strauss G, Fuchs G (1993). "Enzymes of a novel autotrophic CO2 fixation pathway in the phototrophic bacterium Chloroflexus aurantiacus, the 3-hydroxypropionate cycle". Eur J Biochem. 215 (3): 633–43. PMID 8354269. डीओआइ:10.1111/j.1432-1033.1993.tb18074.x.
- ↑ Wood H (1991). "Life with CO or CO2 and H2 as a source of carbon and energy". FASEB J. 5 (2): 156–63. PMID 1900793. मूल से 5 अगस्त 2007 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Shively J, van Keulen G, Meijer W (1998). "Something from almost nothing: carbon dioxide fixation in chemoautotrophs". Annu Rev Microbiol. 52: 191–230. PMID 9891798. डीओआइ:10.1146/annurev.micro.52.1.191.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Boiteux A, Hess B (1981). "Design of glycolysis". Philos Trans R Soc Lond B Biol Sci. 293 (1063): 5–22. PMID 6115423. डीओआइ:10.1098/rstb.1981.0056.
- ↑ Pilkis S, el-Maghrabi M, Claus T (1990). "Fructose-2,6-bisphosphate in control of hepatic gluconeogenesis. From metabolites to molecular genetics". Diabetes Care. 13 (6): 582–99. PMID 2162755. डीओआइ:10.2337/diacare.13.6.582.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ अ आ Ensign S (2006). "Revisiting the glyoxylate cycle: alternate pathways for microbial acetate assimilation". Mol Microbiol. 61 (2): 274–6. PMID 16856935. डीओआइ:10.1111/j.1365-2958.2006.05247.x.
- ↑ Finn P, Dice J (2006). "Proteolytic and lipolytic responses to starvation". Nutrition. 22 (7–8): 830–44. PMID 16815497. डीओआइ:10.1016/j.nut.2006.04.008.
- ↑ अ आ Kornberg H, Krebs H (1957). "Synthesis of cell constituents from C2-units by a modified tricarboxylic acid cycle". Nature. 179 (4568): 988–91. PMID 13430766. डीओआइ:10.1038/179988a0.
- ↑ Rademacher T, Parekh R, Dwek R (1988). "Glycobiology". Annu Rev Biochem. 57: 785–838. PMID 3052290. डीओआइ:10.1146/annurev.bi.57.070188.004033.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Opdenakker G, Rudd P, Ponting C, Dwek R (1993). "Concepts and principles of glycobiology". FASEB J. 7 (14): 1330–7. PMID 8224606. मूल से 18 जून 2007 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ McConville M, Menon A (2000). "Recent developments in the cell biology and biochemistry of glycosylphosphatidylinositol lipids (review)". Mol Membr Biol. 17 (1): 1–16. PMID 10824734. डीओआइ:10.1080/096876800294443.
- ↑ Chirala S, Wakil S (2004). "Structure and function of animal fatty acid synthase". Lipids. 39 (11): 1045–53. PMID 15726818. डीओआइ:10.1007/s11745-004-1329-9.
- ↑ White S, Zheng J, Zhang Y (2005). "The structural biology of type II fatty acid biosynthesis". Annu Rev Biochem. 74: 791–831. PMID 15952903. डीओआइ:10.1146/annurev.biochem.74.082803.133524.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Ohlrogge J, Jaworski J (1997). "Regulation of fatty acid synthesis". Annu Rev Plant Physiol Plant Mol Biol. 48: 109–136. PMID 15012259. डीओआइ:10.1146/annurev.arplant.48.1.109.
- ↑ Dubey V, Bhalla R, Luthra R (2003). "An overview of the non-mevalonate pathway for terpenoid biosynthesis in plants" (PDF). J Biosci. 28 (5): 637–46. PMID 14517367. डीओआइ:10.1007/BF02703339. मूल से 15 अप्रैल 2007 को पुरालेखित (PDF). अभिगमन तिथि 27 अगस्त 2010.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ अ आ Kuzuyama T, Seto H (2003). "Diversity of the biosynthesis of the isoprene units". Nat Prod Rep. 20 (2): 171–83. PMID 12735695. डीओआइ:10.1039/b109860h.
- ↑ Grochowski L, Xu H, White R (2006). "Methanocaldococcus jannaschii uses a modified mevalonate pathway for biosynthesis of isopentenyl diphosphate". J Bacteriol. 188 (9): 3192–8. PMID 16621811. डीओआइ:10.1128/JB.188.9.3192-3198.2006. पी॰एम॰सी॰ 1447442. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Lichtenthaler H (1999). "The 1-Ddeoxy-D-xylulose-5-phosphate pathway of isoprenoid biosynthesis in plants". Annu Rev Plant Physiol Plant Mol Biol. 50: 47–65. PMID 15012203. डीओआइ:10.1146/annurev.arplant.50.1.47.
- ↑ अ आ Schroepfer G (1981). "Sterol biosynthesis". Annu Rev Biochem. 50: 585–621. PMID 7023367. डीओआइ:10.1146/annurev.bi.50.070181.003101.
- ↑ Lees N, Skaggs B, Kirsch D, Bard M (1995). "Cloning of the late genes in the ergosterol biosynthetic pathway of Saccharomyces cerevisiae—a review". Lipids. 30 (3): 221–6. PMID 7791529. डीओआइ:10.1007/BF02537824.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Guyton, Arthur C.; John E. Hall (2006). Textbook of Medical Physiology. Philadelphia: Elsevier. पपृ॰ 855–6. आई॰ऍस॰बी॰ऍन॰ 0-7216-0240-1.
- ↑ Ibba M, Söll D (2001). "The renaissance of aminoacyl-tRNA synthesis". EMBO Rep. 2 (5): 382–7. PMID 11375928.
- ↑ Lengyel P, Söll D (1969). "Mechanism of protein biosynthesis". Bacteriol Rev. 33 (2): 264–301. PMID 4896351. पी॰एम॰सी॰ 378322.
- ↑ अ आ Rudolph F (1994). "The biochemistry and physiology of nucleotides". J Nutr. 124 (1 Suppl): 124S–127S. PMID 8283301. Zrenner R, Stitt M, Sonnewald U, Boldt R (2006). "Pyrimidine and purine biosynthesis and degradation in plants". Annu Rev Plant Biol. 57: 805–36. PMID 16669783. डीओआइ:10.1146/annurev.arplant.57.032905.105421.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Stasolla C, Katahira R, Thorpe T, Ashihara H (2003). "Purine and pyrimidine nucleotide metabolism in higher plants". J Plant Physiol. 160 (11): 1271–95. PMID 14658380. डीओआइ:10.1078/0176-1617-01169.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Smith J (1995). "Enzymes of nucleotide synthesis". Curr Opin Struct Biol. 5 (6): 752–7. PMID 8749362. डीओआइ:10.1016/0959-440X(95)80007-7.
- ↑ Testa B, Krämer S (2006). "The biochemistry of drug metabolism—an introduction: part 1. Principles and overview". Chem Biodivers. 3 (10): 1053–101. PMID 17193224. डीओआइ:10.1002/cbdv.200690111.
- ↑ Danielson P (2002). "The cytochrome P450 superfamily: biochemistry, evolution and drug metabolism in humans". Curr Drug Metab. 3 (6): 561–97. PMID 12369887. डीओआइ:10.2174/1389200023337054.
- ↑ King C, Rios G, Green M, Tephly T (2000). "UDP-glucuronosyltransferases". Curr Drug Metab. 1 (2): 143–61. PMID 11465080. डीओआइ:10.2174/1389200003339171.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Sheehan D, Meade G, Foley V, Dowd C (2001). "Structure, function and evolution of glutathione transferases: implications for classification of non-mammalian members of an ancient enzyme superfamily". Biochem J. 360 (Pt 1): 1–16. PMID 11695986. डीओआइ:10.1042/0264-6021:3600001. पी॰एम॰सी॰ 1222196. मूल से 20 जून 2002 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Galvão T, Mohn W, de Lorenzo V (2005). "Exploring the microbial biodegradation and biotransformation gene pool". Trends Biotechnol. 23 (10): 497–506. PMID 16125262. डीओआइ:10.1016/j.tibtech.2005.08.002.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Janssen D, Dinkla I, Poelarends G, Terpstra P (2005). "Bacterial degradation of xenobiotic compounds: evolution and distribution of novel enzyme activities". Environ Microbiol. 7 (12): 1868–82. PMID 16309386. डीओआइ:10.1111/j.1462-2920.2005.00966.x.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Davies K (1995). "Oxidative stress: the paradox of aerobic life". Biochem Soc Symp. 61: 1–31. PMID 8660387.
- ↑ Tu B, Weissman J (2004). "Oxidative protein folding in eukaryotes: mechanisms and consequences". J Cell Biol. 164 (3): 341–6. PMID 14757749. डीओआइ:10.1083/jcb.200311055. पी॰एम॰सी॰ 2172237. मूल से 8 फ़रवरी 2007 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Sies H (1997). "Oxidative stress: oxidants and antioxidants" (PDF). Exp Physiol. 82 (2): 291–5. PMID 9129943. मूल (PDF) से 25 मार्च 2009 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Vertuani S, Angusti A, Manfredini S (2004). "The antioxidants and pro-antioxidants network: an overview". Curr Pharm Des. 10 (14): 1677–94. PMID 15134565. डीओआइ:10.2174/1381612043384655.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ von Stockar U, Liu J (1999). "Does microbial life always feed on negative entropy? Thermodynamic analysis of microbial growth". Biochim Biophys Acta. 1412 (3): 191–211. PMID 10482783. डीओआइ:10.1016/S0005-2728(99)00065-1.
- ↑ Demirel Y, Sandler S (2002). "Thermodynamics and bioenergetics". Biophys Chem. 97 (2–3): 87–111. PMID 12050002. डीओआइ:10.1016/S0301-4622(02)00069-8.
- ↑ Albert R (2005). "Scale-free networks in cell biology". J Cell Sci. 118 (Pt 21): 4947–57. PMID 16254242. डीओआइ:10.1242/jcs.02714. मूल से 12 अप्रैल 2007 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Brand M (1997). "Regulation analysis of energy metabolism". J Exp Biol. 200 (Pt 2): 193–202. PMID 9050227. मूल से 29 मार्च 2007 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010.
- ↑ Soyer O, Salathé M, Bonhoeffer S (2006). "Signal transduction networks: topology, response and biochemical processes". J Theor Biol. 238 (2): 416–25. PMID 16045939. डीओआइ:10.1016/j.jtbi.2005.05.030.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ अ आ Salter M, Knowles R, Pogson C (1994). "Metabolic control". Essays Biochem. 28: 1–12. PMID 7925313.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Westerhoff H, Groen A, Wanders R (1984). "Modern theories of metabolic control and their applications (review)". Biosci Rep. 4 (1): 1–22. PMID 6365197. डीओआइ:10.1007/BF01120819.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Fell D, Thomas S (1995). "Physiological control of metabolic flux: the requirement for multisite modulation". Biochem J. 311 (Pt 1): 35–9. PMID 7575476. पी॰एम॰सी॰ 1136115.
- ↑ Hendrickson W (2005). "Transduction of biochemical signals across cell membranes". Q Rev Biophys. 38 (4): 321–30. PMID 16600054. डीओआइ:10.1017/S0033583506004136.
- ↑ Cohen P (2000). "The regulation of protein function by multisite phosphorylation—a 25 year update". Trends Biochem Sci. 25 (12): 596–601. PMID 11116185. डीओआइ:10.1016/S0968-0004(00)01712-6.
- ↑ Lienhard G, Slot J, James D, Mueckler M (1992). "How cells absorb glucose". Sci Am. 266 (1): 86–91. PMID 1734513. डीओआइ:10.1038/scientificamerican0192-86.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Roach P (2002). "Glycogen and its metabolism". Curr Mol Med. 2 (2): 101–20. PMID 11949930. डीओआइ:10.2174/1566524024605761.
- ↑ Newgard C, Brady M, O'Doherty R, Saltiel A (2000). "Organizing glucose disposal: emerging roles of the glycogen targeting subunits of protein phosphatase-1" (PDF). Diabetes. 49 (12): 1967–77. PMID 11117996. डीओआइ:10.2337/diabetes.49.12.1967. मूल से 19 जून 2007 को पुरालेखित (PDF). अभिगमन तिथि 27 अगस्त 2010.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Romano A, Conway T (1996). "Evolution of carbohydrate metabolic pathways". Res Microbiol. 147 (6–7): 448–55. PMID 9084754. डीओआइ:10.1016/0923-2508(96)83998-2.
- ↑ Koch A (1998). "How did bacteria come to be?". Adv Microb Physiol. 40: 353–99. PMID 9889982. डीओआइ:10.1016/S0065-2911(08)60135-6.
- ↑ Ouzounis C, Kyrpides N (1996). "The emergence of major cellular processes in evolution". FEBS Lett. 390 (2): 119–23. PMID 8706840. डीओआइ:10.1016/0014-5793(96)00631-X.
- ↑ Caetano-Anolles G, Kim HS, Mittenthal JE (2007). "The origin of modern metabolic networks inferred from phylogenomic analysis of protein architecture". Proc Natl Acad Sci USA. 104 (22): 9358–63. PMID 17517598. डीओआइ:10.1073/pnas.0701214104. पी॰एम॰सी॰ 1890499.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Schmidt S, Sunyaev S, Bork P, Dandekar T (2003). "Metabolites: a helping hand for pathway evolution?". Trends Biochem Sci. 28 (6): 336–41. PMID 12826406. डीओआइ:10.1016/S0968-0004(03)00114-2.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Light S, Kraulis P (2004). "Network analysis of metabolic enzyme evolution in Escherichia coli". BMC Bioinformatics. 5: 15. PMID 15113413. डीओआइ:10.1186/1471-2105-5-15. पी॰एम॰सी॰ 394313. Alves R, Chaleil R, Sternberg M (2002). "Evolution of enzymes in metabolism: a network perspective". J Mol Biol. 320 (4): 751–70. PMID 12095253. डीओआइ:10.1016/S0022-2836(02)00546-6.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Kim HS, Mittenthal JE, Caetano-Anolles G (2006). "MANET: tracing evolution of protein architecture in metabolic networks". BMC Bioinformatics. 19 (7): 351. PMID 16854231. डीओआइ:10.1186/1471-2105-7-351. पी॰एम॰सी॰ 1559654.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Teichmann SA, Rison SC, Thornton JM, Riley M, Gough J, Chothia C (2001). "Small-molecule metabolsim: an enzyme mosaic". Trends Biotechnol. 19 (12): 482–6. PMID 11711174. डीओआइ:10.1016/S0167-7799(01)01813-3.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Spirin V, Gelfand M, Mironov A, Mirny L (2006). "A metabolic network in the evolutionary context: multiscale structure and modularity". Proc Natl Acad Sci USA. 103 (23): 8774–9. PMID 16731630. डीओआइ:10.1073/pnas.0510258103. पी॰एम॰सी॰ 1482654. मूल से 12 सितंबर 2019 को पुरालेखित. अभिगमन तिथि 27 अगस्त 2010. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Lawrence J (2005). "Common themes in the genome strategies of pathogens". Curr Opin Genet Dev. 15 (6): 584–8. PMID 16188434. डीओआइ:10.1016/j.gde.2005.09.007. Wernegreen J (2005). "For better or worse: genomic consequences of intracellular mutualism and parasitism". Curr Opin Genet Dev. 15 (6): 572–83. PMID 16230003. डीओआइ:10.1016/j.gde.2005.09.013.
- ↑ Pál C, Papp B, Lercher M, Csermely P, Oliver S, Hurst L (2006). "Chance and necessity in the evolution of minimal metabolic networks". Nature. 440 (7084): 667–70. PMID 16572170. डीओआइ:10.1038/nature04568.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Rennie M (1999). "An introduction to the use of tracers in nutrition and metabolism". Proc Nutr Soc. 58 (4): 935–44. PMID 10817161. डीओआइ:10.1017/S002966519900124X.
- ↑ Phair R (1997). "Development of kinetic models in the nonlinear world of molecular cell biology". Metabolism. 46 (12): 1489–95. PMID 9439549. डीओआइ:10.1016/S0026-0495(97)90154-2.
- ↑ Sterck L, Rombauts S, Vandepoele K, Rouzé P, Van de Peer Y (2007). "How many genes are there in plants (... and why are they there)?". Curr Opin Plant Biol. 10 (2): 199–203. PMID 17289424. डीओआइ:10.1016/j.pbi.2007.01.004.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Borodina I, Nielsen J (2005). "From genomes to in silico cells via metabolic networks". Curr Opin Biotechnol. 16 (3): 350–5. PMID 15961036. डीओआइ:10.1016/j.copbio.2005.04.008.
- ↑ Gianchandani E, Brautigan D, Papin J (2006). "Systems analyses characterize integrated functions of biochemical networks". Trends Biochem Sci. 31 (5): 284–91. PMID 16616498. डीओआइ:10.1016/j.tibs.2006.03.007.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Duarte NC, Becker SA, Jamshidi N; एवं अन्य (2007). "Global reconstruction of the human metabolic network based on genomic and bibliomic data". Proc. Natl. Acad. Sci. U.S.A. 104 (6): 1777–82. PMID 17267599. डीओआइ:10.1073/pnas.0610772104. पी॰एम॰सी॰ 1794290. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद); Explicit use of et al. in:|author=(मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Goh KI, Cusick ME, Valle D, Childs B, Vidal M, Barabási AL (2007). "The human disease network". Proc. Natl. Acad. Sci. U.S.A. 104 (21): 8685–90. PMID 17502601. डीओआइ:10.1073/pnas.0701361104. पी॰एम॰सी॰ 1885563. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Lee DS, Park J, Kay KA, Christakis NA, Oltvai ZN, Barabási AL (2008). "The implications of human metabolic network topology for disease comorbidity". Proc. Natl. Acad. Sci. U.S.A. 105 (29): 9880–9885. PMID 18599447. डीओआइ:10.1073/pnas.0802208105. पी॰एम॰सी॰ 2481357. नामालूम प्राचल
|month=की उपेक्षा की गयी (मदद)सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) - ↑ Csete M, Doyle J (2004). "Bow ties, metabolism and disease". Trends Biotechnol. 22 (9): 446–50. PMID 5249808. डीओआइ:10.1016/j.tibtech.2004.07.007. पी॰एम॰सी॰ 225248.
- ↑ Ma HW, Zeng AP (2003). "The connectivity structure, giant strong component and centrality of metabolic networks". Bioinformatics. 19 (11): 1423–30. PMID 12874056. डीओआइ:10.1093/bioinformatics/btg177.
- ↑ Zhao J, Yu H, Luo JH, Cao ZW, Li YX (2006). "Hierarchical modularity of nested bow-ties in metabolic networks". BMC Bioinformatics. 7: 386. PMID 16916470. डीओआइ:10.1186/1471-2105-7-386. पी॰एम॰सी॰ 1560398.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Thykaer J, Nielsen J (2003). "Metabolic engineering of beta-lactam production". Metab Eng. 5 (1): 56–69. PMID 12749845. डीओआइ:10.1016/S1096-7176(03)00003-X. González-Pajuelo M, Meynial-Salles I, Mendes F, Andrade J, Vasconcelos I, Soucaille P (2005). "Metabolic engineering of Clostridium acetobutylicum for the industrial production of 1,3-propanediol from glycerol". Metab Eng. 7 (5–6): 329–36. PMID 16095939. डीओआइ:10.1016/j.ymben.2005.06.001.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link) Krämer M, Bongaerts J, Bovenberg R, Kremer S, Müller U, Orf S, Wubbolts M, Raeven L (2003). "Metabolic engineering for microbial production of shikimic acid". Metab Eng. 5 (4): 277–83. PMID 14642355. डीओआइ:10.1016/j.ymben.2003.09.001.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ Koffas M, Roberge C, Lee K, Stephanopoulos G (1999). "Metabolic engineering". Annu Rev Biomed Eng. 1: 535–57. PMID 11701499. डीओआइ:10.1146/annurev.bioeng.1.1.535.सीएस1 रखरखाव: एक से अधिक नाम: authors list (link)
- ↑ "Metabolism". The Online Etymology Dictionary. मूल से 1 अक्तूबर 2010 को पुरालेखित. अभिगमन तिथि 2007-02-20.
- ↑ डॉ॰ अबू शादी अल-रौबी (1982), "इब्न अल-नफीस एज़ अ फीलॉज़ोफर", सिमपोज़ियम ऑन इब्न अल नफीस, सेकण्ड इंटरनेशनल कांफेरेंस ऑन इस्लामिक मेडिसिन: इस्लामिक मेडिकल ओर्गानैज़ेशन, कोवैत (सीएफ. इब्नुल-नफीस एस अ फिलोज़फर, इनसैक्लोपिडिया ऑफ़ इस्लामिक वर्ल्ड [1]).
- ↑ Eknoyan G (1999). "Santorio Sanctorius (1561–1636) - founding father of metabolic balance studies". Am J Nephrol. 19 (2): 226–33. PMID 10213823. डीओआइ:10.1159/000013455.
- ↑ विलियम्स, एच. एस. (1904) अ हिस्टरी ऑफ़ साइंस: इन फाइव वोल्युम्स Archived 2012-05-09 at the वेबैक मशीन.वोल्यूम IV: मॉडर्न डेवेलपमेंट ऑफ़ द क्लिनिकल एंड बायोलॉजिकल साइंसेस हार्पर एंड ब्रदर्स Archived 2012-05-09 at the वेबैक मशीन (न्यू यॉर्क) 26-03-2007 में पुनःप्राप्त
- ↑ Dubos J. (1951). "Louis Pasteur: Free Lance of Science, Gollancz. Quoted in Manchester K. L. (1995) Louis Pasteur (1822–1895)—chance and the prepared mind". Trends Biotechnol. 13 (12): 511–515. PMID 8595136. डीओआइ:10.1016/S0167-7799(00)89014-9.
- ↑ Kinne-Saffran E, Kinne R (1999). "Vitalism and synthesis of urea. From Friedrich Wöhler to Hans A. Krebs". Am J Nephrol. 19 (2): 290–4. PMID 10213830. डीओआइ:10.1159/000013463.
- ↑ एडुअर्ड बकनर्स 1907 नोबल लेक्चर Archived 2017-07-08 at the वेबैक मशीन एट http://nobelprize.ओर्ग[मृत कड़ियाँ] 20-03-2007 से पुनःप्राप्त
- ↑ Kornberg H (2000). "Krebs and his trinity of cycles". Nat Rev Mol Cell Biol. 1 (3): 225–8. PMID 11252898. डीओआइ:10.1038/35043073.
- ↑ Krebs HA, Henseleit K (1932). "Untersuchungen über die Harnstoffbildung im tierkorper". Z. Physiol. Chem. 210: 33–66.
Krebs H, Johnson W (1937). "Metabolism of ketonic acids in animal tissues". Biochem J. 31 (4): 645–60. PMID 16746382. पी॰एम॰सी॰ 1266984. नामालूम प्राचल|month=की उपेक्षा की गयी (मदद)
आगे पढ़ें[संपादित करें]
परिचयात्मक
- Rose, S. और Mileusnic, R., द कैमिस्ट्री ऑफ़ लाइफ . (पेंगुइन प्रेस विज्ञान, 1999), आईएसबीएन (ISBN) 0-14027-273-9
- Schneider, E. D. और Sagan, D., इनटू द कूल: एनेर्जी फ्लो, थर्मोडैनामिक्स, एंड लाइफ . (शिकागो विश्वविद्यालय का प्रेस, 2005), आईएसबीएन (ISBN) 0-22673-936-8
- Lane, N., ऑक्सीजन: द मॉलीक्यूल डैट मेड द वर्ल्ड . (ऑक्सफोर्ड यूनिवर्सिटी प्रेस, अमरीका, 2004), ISBN 0-19-860783-0
प्रगतिशील
- Price, N. और Stevens, L., फंडामेंटल्स ऑफ़ एनज़ैमोलॉजी: सेल एंड मॉलीक्युलर बैओलॉजी ऑफ़ कटालिटिक प्रोटीन . (ऑक्सफोर्ड यूनिवर्सिटी प्रेस, 1999), ISBN 0-19-850229-X
- Berg, J. Tymoczko, J. और Stryer, L., जैव रसायन (डब्ल्यू.एच फ्रीमैन और कंपनी, 2002), आईएसबीएन (ISBN 0-7167-4955-6)
- Cox, M. और Nelson, D. L., लेहनिंगर प्रिंसिपल्स ऑफ़ बायोकेमिस्ट्री . (पलग्रेव मैकमिलन, 2004), आईएसबीएन (ISBN) 0-71674-339-6
- Brock, T. D. Madigan, M. T. Martinko, J. और Parker J., ब्रोक्स बायोलॉजी ऑफ़ मैक्रोऔरगेनिस्म . (बेंजामिन कम्मिंग्स, 2002), आईएसबीएन {ISBN} 0-13066-271-2
- Da Silva, J.J.R.F. और Williams, R. J. P., द बायोलॉजिकल केमिस्ट्री ऑफ़ द एलिमेंट्स: द इनओर्गानिक केमिस्ट्री ऑफ़ लाइफ . (क्लारेंडन प्रेस, 1991), आईएसबीएन (ISBN) 0-19855-598-9
- Nicholls, D. G. और Ferguson, S. J., बायोएनेर्जेटिक्स . (एकाडेमिक प्रेस इंक, 2002), आईएसबीएन (ISBN) 0-12518-121-3
बाहरी लिंक्स[संपादित करें]
- मेटाबॉलिज़्म, सेललियुलर रेसपिरेशन एंड फॉटोसेंथेसिस द वरचुएल लाइबरेरी ऑफ़ बायोकेमिस्ट्री एंड सेल बायोलॉजी एट biochemweb.org
- मेटाबॉलिज़्म की बायोकेमिस्ट्री
- एडवांस्ड एनीमल मेटाबॉलिज़्म कैलकूलेटर्स/ इंटरएक्टिव लर्निंग टूल्स
- माइक्रोबियल मेटाबॉलिज़्म सरल सिंहावलोकन. स्कूल स्तर.
- बायोकेमिस्ट्री रास्ते मेटाबोलिक प्रमुख चयापचय रास्ते का ग्राफिकल प्रतिनिधित्व.
- बायोलॉजिस्ट्स के लिए रसायन विज्ञान चयापचय की रसायन विज्ञान का परिचय. स्कूल स्तर.
- स्पार्कनेस एसएटी (SAT) बायोकेमिस्ट्री जैव रसायन का अवलोकन. स्कूल स्तर.
- एमआईटी (MIT) बायोलॉजी हाईपरटेक्स्टबूक आणविक जीव-विज्ञान के अंडरग्रेजुएट-स्तर के मार्गदर्शन.
- टॉपिक्स इन मेडिकल बायोकेमिस्ट्री गाइड टू हिउमन मेटाबॉलिक पाथवेज़. स्कूल स्तर.
- द मेडिकल बायोकेमिस्ट्री पेज मानव चयापचय पर व्यापक संसाधन.
- इंटरएक्टिव फ्लो चार्ट ऑफ़ द मेजर मेटाबॉलिक रास्ते का फ्लो चार्ट
- मेटाबॉलिज़्म रेफेरेंस पाथवेज़
- गाइड टू ग्लाईकोलिसिस स्कूल स्तर पर.
- वेबैक मशीन पर The Nitrogen cycle and Nitrogen fixation at the Wayback Machine.
- डाउनलोडेबल गाइड टू फॉटोसिंथेसिस स्कूल स्तर.
- फॉटोसिंथेसिस क्या है? प्रकाश संश्लेषण लेख और संसाधनों का संग्रह.
जैवरासायनिक परिवार: कार्बोहाइड्रेट (ग्लाइकोसाइड, अल्कोहॉल) · लिपिड (Steroids, Phospholipids, Glycolipids, वसीय अम्ल, Tetrapyrroles) · प्रोटीन (अमीनो अम्ल, पेप्टाइड, Glycoproteins) · Nucleobases/Nucleic acids
